CRISPR-Cas9转录激活文库
CRISPR-Cas9系统是目前最高效,精准,简单易操作的基因编辑系统。最近,CRISPR-Cas9也被应用于基因组水平的功能获得型(gain-of-function,GOF)遗传筛选,即CRISPRa (CRISPR activation)。其基本原理是:通过融合催化活性失活的效应蛋白Cas9(dCas9)和转录激活调控元件如VP64 或 p65的转录激活域,在sgRNA的引导下,靶向目标基因启动子区,激活目标基因的转录【1】。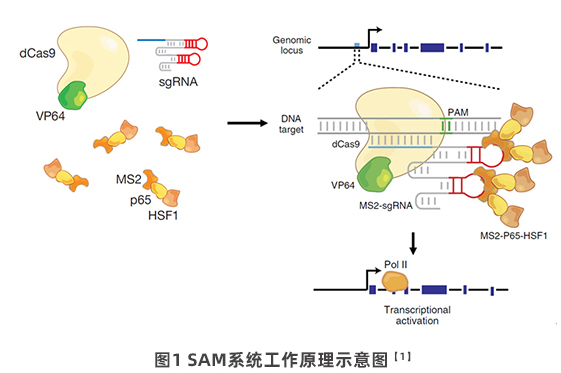
安升达公司提供Broad授权的CRISPR-Cas9 synergistic activation mediator (SAM) 系统【3】。研究发现,在不同的细胞系和物种中对比其他CRISPRa系统如SunTag(通过表位的重复肽阵列与单链可变片段抗体配对来募集多个VP64激活域)和VPR(dCas9融合三种转录激活域即VP64-p65-Rta),SAM在某些情况下可诱导更有效的激活【4】。
SAM系统包含三部分组件:
1,细胞核定位的催化活性缺失dCas9与VP64激活域的融合蛋白,NLS-dCas9-VP64。
2,修改的sgRNA骨架,在四环区(tetraloop)和茎环2区(stem loop 2)加入两个发卡结构适配子(hairpin aptamer),可以结合二聚化的MS2噬菌体外壳蛋白。
3,MS2与p65和人类热激转录因子HSF1转录激活域的融合蛋白,MS2-p65-HSF1。
如图1所示,sgRNA与dCas9组装成复合体,发卡结构结合MS2蛋白,最终将VP64,p65和HSF1转录激活域招募到基因组的特定位置如启动子序列,转录起始位点(Transcription start site, TSS)附近,提高目标基因的转录水平。
CRISPR-Cas9 SAM筛选详细流程
CRISPR-Cas9 SAM系统有三部分,因此需要先构建稳定表达SAM系统的细胞系。以MOI<1将NLS-dCas9-VP64 和MS2-p65-HSF1慢病毒转染目标细胞(此步骤也可以将sgRNA表达框与dCas9-VP64构建在同一个载体骨架中,则只需先构建稳定表达MS2-p65-HSF1的细胞系),进行抗生素筛选,筛选完成后得到稳定表达dCas9-VP64 和MS2-p65-HSF1的细胞系,以MOI≈0.3将SAM sgRNA慢病毒文库转染上述筛选完成的细胞,抗生素筛选后,达到约每个sgRNA有500个细胞覆盖;根据实验目的进行小分子,药物或其他条件处理后,分离DNA,扩增DNA,NGS建库测序,数据分析(图2)。
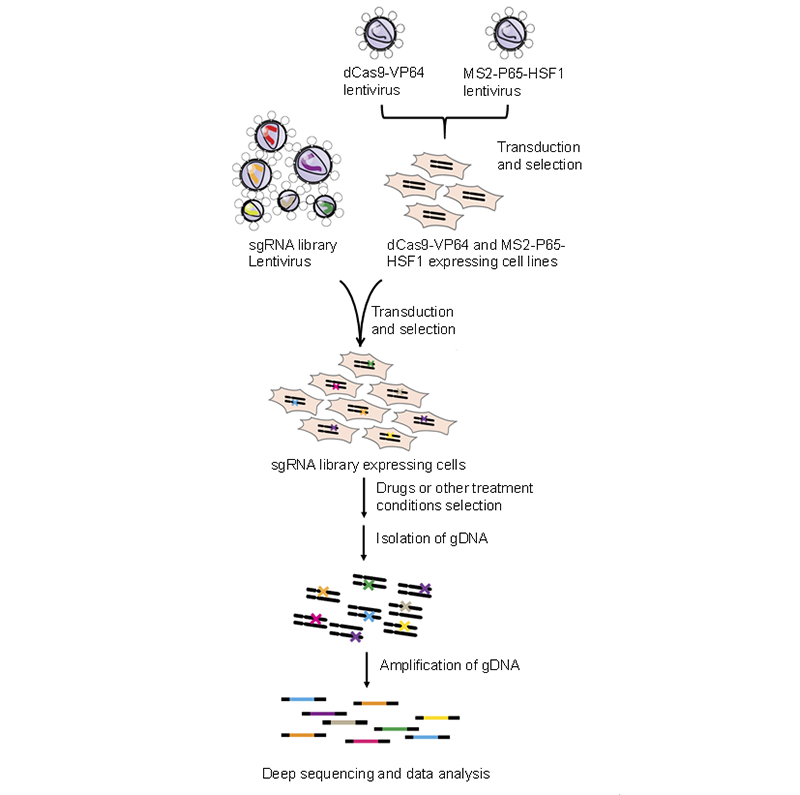
图2 CRISPR-Cas9 SAM文库筛选流程示意图【1】
注意事项:包装完成的sgRNA慢病毒文库以比较低的MOI如0.3转染目标细胞,经过抗生素筛选,尽量使每个细胞仅接受一个拷贝的sgRNA,即每个细胞仅有一个目标基因被过表达;转染细胞的数量建议每条sgRNA>500个细胞。
CRISPR-Cas9转录激活文库应用案例
1. 药物作用机制的研究:Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex
在顽固的疾病治疗中,常常有病人对治疗药物产生先天或后天的抗药性。因此,在基因组水平理解哪些基因会影响药物的功效能够为临床治疗提供更准确的方案。Konermann等【3】应用SAM转录激活文库在A375(BRAF(V600E))黑色素瘤细胞中筛选抵抗黑色素瘤药物(BRAF inhibitor,PLX-4720)的基因位点。该研究构建了基因组水平的SAM sgRNA慢病毒文库,包含70290个sgRNAs,靶定RefSeq数据库中23430个coding isoforms转录起始位点上游200 bp位置,每个基因设计3个sgRNAs。通过文库筛选,NGS分析和验证,最终鉴定到一系列已知的和新的参与PLX-4720抗药性相关的候选基因(图3)。
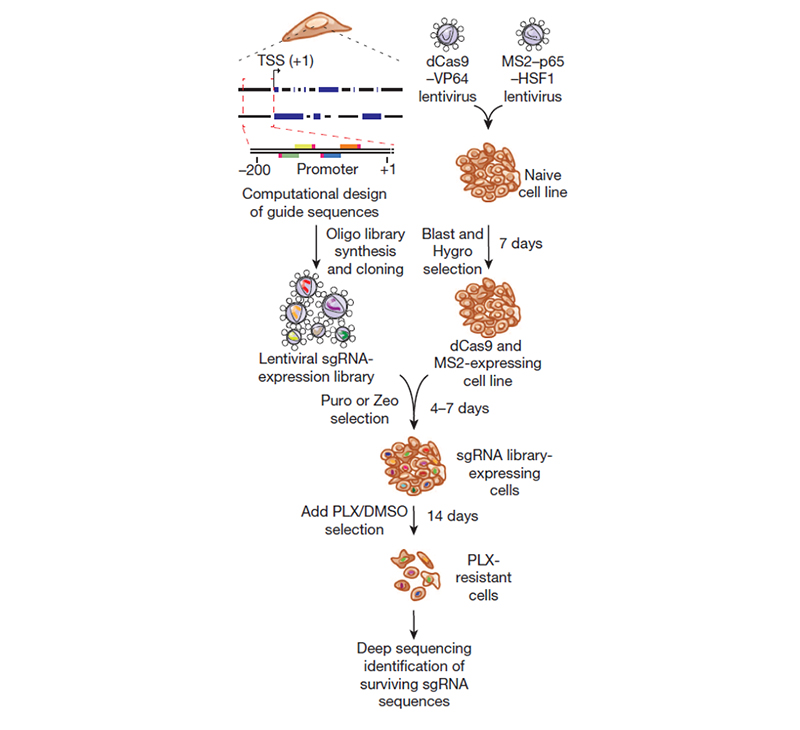
图3 SAM转录激活系统筛选流程图【3】
2. 长链非编码RNA功能研究:Genome-scale activation screen identifies a lncRNA locus regulating a gene neighborhood
哺乳动物基因组包含成千上万个可以转录长的非编码RNA(long noncoding RNAs,lncRNA)的基因座,其中一些已知在多种细胞过程中起关键调控作用。尽管lncRNAs可能以反式(in trans, act non-locally)的方式起作用,但也有证据表明,许多lncRNAs以顺势(in cis, act locally)的方式参与调控,即该基因座的lncRNA会对附近基因的表达起调控作用。然而,直到目前,对lncRNA功能和机制的研究仍充满挑战。为了探索该问题,Joung等【5】构建了基因组水平上靶定10000多个lncRNA转录起始位点的sgRNA慢病毒文库,并利用SAM转录激活系统在黑色素瘤细胞中筛选鉴定到11个新的介导BRAF抑制剂(vemurafenib)抗性的lncRNA基因座。对其中一个候选基因:EMICERI 进行详细功能分析发现,该lncRNA的转录激活导致四个相邻蛋白质编码基因的剂量依赖性激活,并且其中一个蛋白编码基因导致了BRAF抑制剂抗性表型。该研究为系统的探索非编码RNA的功能提供了参考。
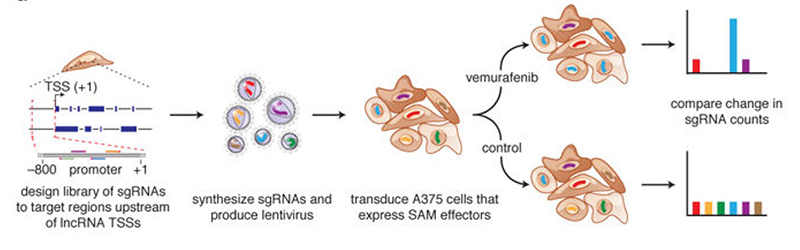
图4 SAM转录激活系统鉴定参与抗vemurafenib的lncRNAs【5】
安升达CRISPRa产品介绍
sgRNA文库构建及质检的优势:
安升达定制化sgRNA文库质量标准
项目经验
| 文库编号 | 文库1 | 文库2 | 文库3 |
| 平均测序深度 | 1015 | 420 | 444 |
| 均一性 | 2.36 | 2.64 | 2.57 |
| sgRNA 文库容量 | 4997 | 86490 | 281888 |
| 覆盖度 | 100% | 100% | 99.97% |
案例说明:我们统计了三个不同sgRNA库容量大小的的质量标准分析,分别代表了亚文库,全基因组文库和超大文库三种不同应用场景的文库; NGS测序分析结果表明:安升达构建的不同标准的文库,其文库质量,覆盖度和均一性都达到了较高标准。
文献引用:
[1] Joung, Julia, et al. “Genome-scale CRISPR-Cas9 knockout and transcriptional activation screening.” Nature protocols 12.4 (2017): 828-863.
[2] Cruz, Cristina, et al. “A gain-of-function screen identifying genes required for growth and pattern formation of the Drosophila melanogaster wing.” Genetics 183.3 (2009): 1005-1026.
[3] Konermann, Silvana, et al. “Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex.” Nature 517.7536 (2015): 583-588.
[4] Chavez, Alejandro, et al. “Comparison of Cas9 activators in multiple species.” Nature methods 13.7 (2016): 563-567.
[5] Joung, Julia, et al. “Genome-scale activation screen identifies a lncRNA locus regulating a gene neighbourhood.” Nature 548.7667 (2017): 343-346.
安升达提供的CRISPR/Cas9技术服务的授权来自Broad研究所。