三文读懂PCA和PCoA(一)
在微生物NGS测序领域的高分文章中,PCA(主成分分析)和PCoA(主坐标分析)会很常见。甚至在RNA分析领域,很多研究和文章也会依据基因的表达量作PCA和PCoA分析。
常见的PCA和PCoA分析以下图的形式呈现:
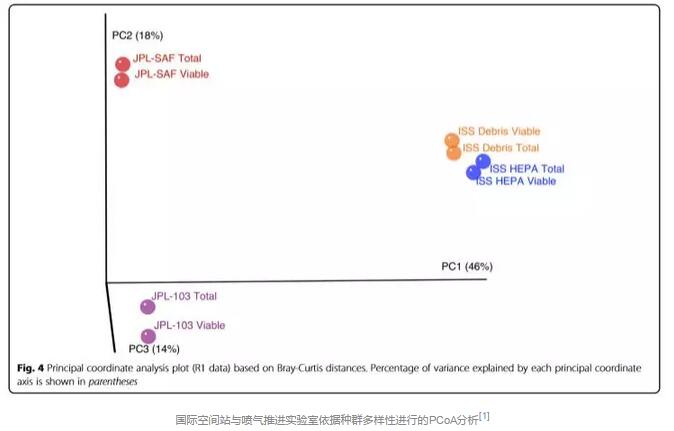
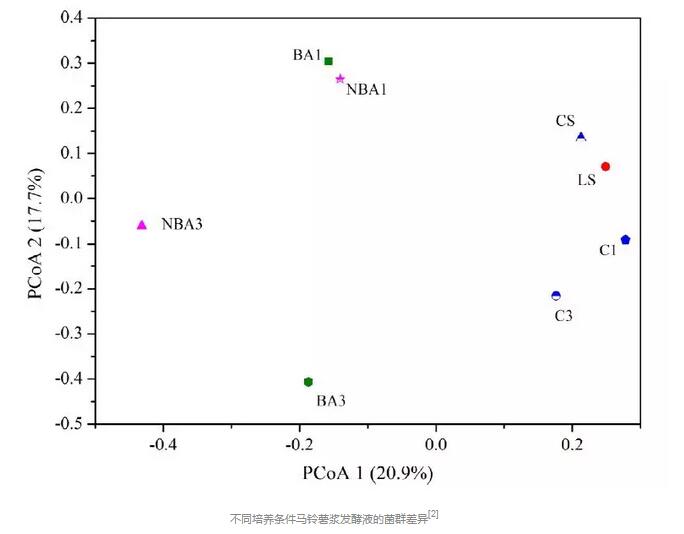
很明显,我们可以通过分析坐标轴中样本和样本之间的距离直观地看到2个样本或2组样本之间的菌群差异性。若2个样本或2组样本之间的直线距离较近,则表示这2个样本或2组样本的菌群差异性较小;相反,若2个样本或2组样本之间的直线距离较远,则表示它们之间菌群差异性较大。所以,PCA和PCoA所呈现的结果,具有直观性(直接看两点之间的距离)和完整性(呈现所有样本),且数据易于分析和解读(大家都看得懂)。
那么,PCA和PCoA是如何定义的?PCA和PCoA之间是否有区别?何时该选用PCA或何时该选用PCoA?PCA和PCoA背后的分析原理如何?相信这些问题是比较困扰读者的。
PCA和PCoA的定义
PCA(Principal Components Analysis)即主成分分析,也称主分量分析或主成分回归分析法,首先利用线性变换,将数据变换到一个新的坐标系统中;然后再利用降维的思想,使得任何数据投影的第一大方差在第一个坐标(称为第一主成分)上,第二大方差在第二个坐标(第二主成分)上。这种降维的思想首先减少数据集的维数,同时还保持数据集的对方差贡献最大的特征,最终使数据直观呈现在二维坐标系。
PCoA(Principal Co-ordinates Analysis)分析即主坐标分析,可呈现研究数据相似性或差异性的可视化坐标,是一种非约束性的数据降维分析方法,可用来研究样本群落组成的相似性或相异性。它与PCA类似,通过一系列的特征值和特征向量进行排序后,选择主要排在前几位的特征值,找到距离矩阵中最主要的坐标,结果是数据矩阵的一个旋转,它没有改变样本点之间的相互位置关系,只是改变了坐标系统。两者的区别为PCA是基于样本的相似系数矩阵(如欧式距离)来寻找主成分,而PCoA是基于距离矩阵(欧式距离以外的其他距离)来寻找主坐标。
好吧,定义比较抽象,我们还是无法看懂看透PCA和PCoA。不急,下面的文字很重要~~~
PCA和PCoA的区别
1.PCA的理解
a. 假如有3个实验样本,它们共有1个物种x,那么我们其实可以用物种x的相对丰度来表示样本和样本之间的差异。这样我们就可以画一个一维坐标轴,将这3个样本的物种x的丰度表示在一维轴线上,如下图所示:
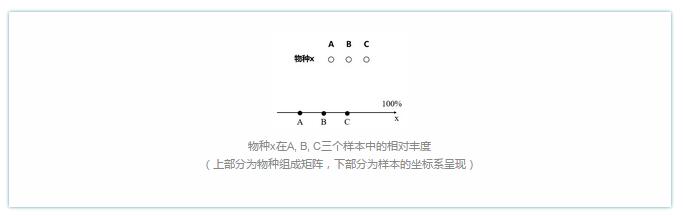
此时数据不发生偏移,样本和样本之间的距离代表样本之间的物种丰度差异(实际上样本A和B间的距离即为A中的物种x的丰度与B中物种x的丰度的差值)。
b. 假如有3个实验样本,它们共有2个物种:x和y。那么我们其实可以用物种x和物种y的相对丰度来在二维坐标系中定位样本。A=(x1,y1), B=(x2,y2),C=(x3,y3),如下图所示:

此时数据不发生偏移,样本和样本之间的距离代表样本之间的物种丰度差异。
c. 假如有3个实验样本,它们共有k个物种: x, y, z…………k。那么我们其实可以用物种x, y, z…………k的丰度来定位样本A=(x1,y1,z1……………k1)。同理,样本B与C也可以用这种形式表示。细心的同学可以发现,其实A=(x1,y1,z1……………k1)是一组向量,而且是k维向量(A=(x1)是一维向量,A=(x1,y1)是二维向量,A=(x1,y1,z1)是三维向量)。但是k维向量无法在二维坐标系(平面)中表示(一维和二维向量可以,如上a和b两种情况)。此时我们要么将K维向量作出一些取舍,如削去一些不重要的向量仅保留2个关键向量(削去一些不重要的物种仅保留2个关键物种);要么将K维向量投射到二维坐标系中(降维),但是此时数据便会损失,例如下图,我们将二维坐标系中的数据投射到一维坐标系中,实际数据会折扣掉一部分(A和B的直线距离为5,投射到x轴的一维距离为4,投射到y轴的一维距离为3。从第一维坐标轴上观察A和B的距离只有4,从第二维坐标轴上观察A和B的距离只有3。)。
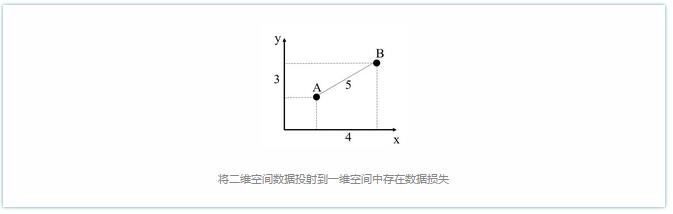
因此将k维空间的数据投射到二维空间上(降维),就会产生数据损失,此时坐标轴的贡献率就不再是100%,而是小于100%(而a和b两种情况无需降维处理,因此贡献率为100%)。此时数据如下图所示:
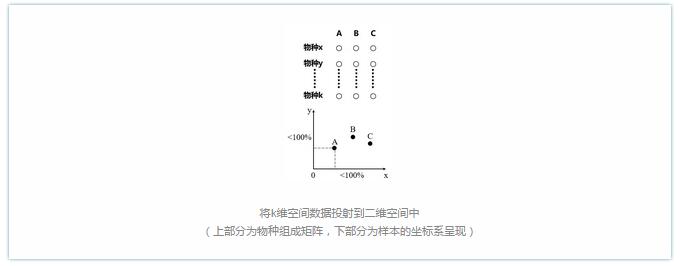
因降维处理,数据发生损失,样本和样本之间的距离代表样本之间的物种丰度差异。
那么如何来选择投影?这就是定义当中所提到的“使得任何数据投影的第一大方差在第一个坐标(称为第一主成分)上,第二大方差在第二个坐标(第二主成分)上”。
2.PCoA的理解
a. 假如有2个实验样本,它们都有很多物种,那么我们可以用Bray-Curtis或UniFrac(或其他算法)计算每个样本的物种组成差异度(用一个数值表示物种相对丰度),数值之间的差异就代表了2个样本的物种相对丰度的差异。这样我们就可以画一个一维坐标轴,将这2个样本表示在一维轴线上,如下图所示:
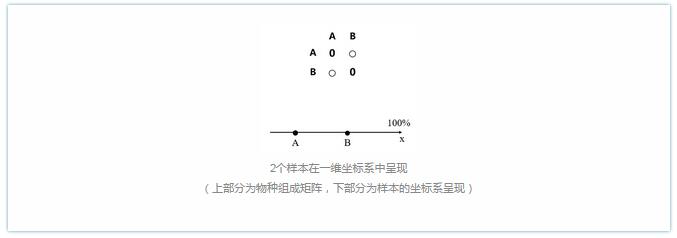
此时数据不发生偏移,样本和样本之间的距离代表样本之间的物种丰度差异。
b. 假如有3个实验样本,同样可以用Bray-Curtis或UniFrac(或其他算法)计算每个样本的物种组成差异度(用一个数值表示物种相对丰度),数值之间的差异就代表了每2个样本的物种相对丰度的差异。这样我们就可以画一个二维坐标轴(三点组成一个面),将这3个样本表示在二维轴线上,如下图所示:
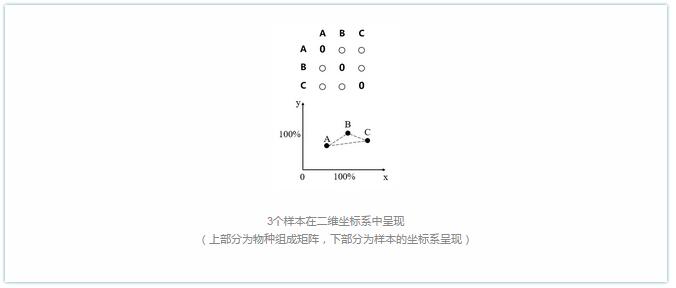
此时数据不发生偏移,样本和样本之间的距离代表样本之间的物种丰度差异。
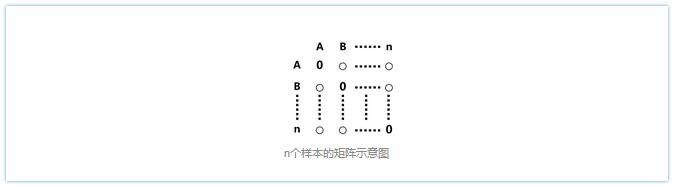
c. 以此类推,假如有n个实验样本,同样可以用Bray-Curtis或UniFrac(或其他算法)计算每个样本的物种组成差异度(用一个数值表示物种相对丰度),数值之间的差异就代表了每2个样本的物种相对丰度的差异。这样我们就可以画一个n-1维坐标轴,将这n个样本表示在n-1维空间中。但是n-1维空间无法在平面上表示(一维和二维除外,三维勉强可以),因此只能利用矩阵呈现,如下图所示:
若要将n-1维的数据在二维坐标系中呈现,需降维处理,即将n-1维的数据投影到二维空间当中,方法与思路同PCA类似。此时,2个坐标轴的贡献率均小于100%,如下图所示:
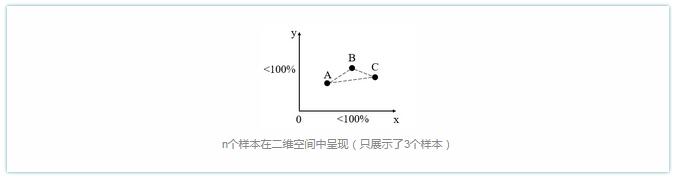
因降维处理,数据发生损失,样本和样本之间的距离代表样本之间的物种丰度差异。
这个时候, PCA和PCoA就好理解了。我们再回过头看定义“PCA是基于样本的相似系数矩阵(如欧式距离)来寻找主成分,而PCoA是基于距离矩阵(欧式距离以外的其他距离)来寻找主坐标”,其实浅显地来理解,就是上面这么回事。
我们知道了PCA和PCoA的定义,也理解了PCA和PCoA的区别,那么它们该何时选用,以及背后的算法如何?欲知后事如何,且听下回分解。
特此声明:
1、 本文仅供读者理解,不涉及专业学术论证;
2、 本文为小编的一点感悟心得,非常欢迎各位业界同行的讨论与交流,同时也非常欢迎各位专家老师的指正,您的一个问题会使我们共同进步!
参考文献:
[1] Aleksandra Checinska et al., Microbiomes of the dust particles collected from the International Space Station and Spacecraft Assembly Facilities. Microbiome. 2015
[2] Zhiman Yang et al., Enhanced methane production via repeated batch bioaugmentation pattern of enriched microbial consortia. Bioresource Technology. 2016
