ssDNA合成
基因组编辑技术CRISPR/Cas9自2013年被《科学》杂志列为年度十大科技进展之一以来,受到越来越多的科研工作者的高度重视,成为基因敲除和基因敲入的主要研究手段。针对在模式生物和细胞中进行基因序列替换、外显子突变位点修复和融合标签等大片段的敲入,现在普遍面临着dsDNA片段的随机插入和片段敲入效率低等风险,难以在精准的基因编辑上被广泛应用。相比之下,ssDNA因其片段敲入效率高、能有效避免dsDNA片段在基因组中随机插入的问题等优势脱颖而出。安升达ssDNA的制备上有丰富的技术经验,目前可以根据客户需求提供150-10k nt的ssDNA产品制备服务, 产品具有正确率高、纯度高、长度长、可以合成绝大部分困难序列等优势,让您的实验后顾无忧。

ssDNA在CRISPR Knock-in中的优势
脱靶效应低
编辑效率高
细胞毒性小
无dsDNA的随机整合
ssDNA制备服务的优势
序列准确率高:单克隆质粒测序验证后制备ssDNA,不经过PCR过程无突变引入;ssDNA Sanger测序验证后发货。
合成长度长:安升达目前可以稳定合成150-10K nt长度的ssDNA。
无dsDNA污染,细胞毒性低:Sanger测序和酶切验证双重保证,确保无dsDNA污染。
合成难度大:安升达目前可以合成大部分难度序列,已成功合成包括ITR序列,大片段正向重复、反向重复等困难序列。
发货规则
| 长度 | 制备量 | 样品发货形式 | 发货文件报告 | 制备周期 |
| 150-500nt | 3µg | 冻干粉常温寄送 |
1.Sanger测序结果(ab1文件) 报告 |
3-4周 |
| 501-2000nt | 3µg | |||
| 10µg | ||||
| 20µg | ||||
| 40µg | ||||
| 2001-5000nt | 3µg | 4-5周 | ||
| 10µg | ||||
| 20µg | ||||
| 40µg | ||||
| 5001-10Knt | 3µg | 5-6周 | ||
| 10µg | ||||
| 20µg | ||||
| 40µg | ||||
| 10Knt以上 | 咨询 | 咨询 |
安升达ssDNA质量介绍
1. Sanger结果
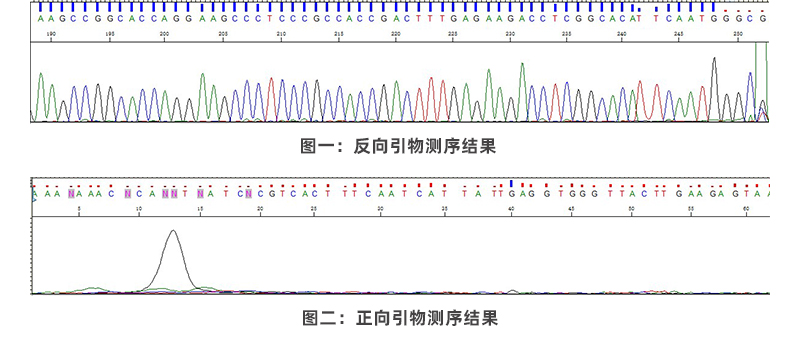
由于ssDNA结构的特殊性,只能用反向测序引物才能测序,正向引物理论上无法和ssDNA结合并测序,安升达会对每一条发货的样品进行反向测序引物测序验证序列100%正确,用正向测序引物测序验证是否由双链残留。
*特殊结构可能导致正向引物也能进行测序,但不是从引物互补的区域开始测序
2. 酶切结果
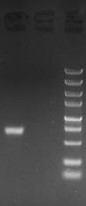
图三:S1核酸酶酶切(泳道一:ssDNA样品;泳道二: ssDNA+S1核酸酶;泳道三:marker)
安升达提供的每一条ssDNA制备样品均经过S1核酸酶酶切验证,确保样品无双链残留(S1核酸酶为特异性降解ssDNA的酶,对dsDNA几乎无活性)
3. S1核酸酶灵敏度测试

图四:S1核酸酶灵敏度测试
从图上可知当样品中含有10ng以上的双链DNA,S1核酸酶无法将之完全降解并可以在凝胶电泳图中显示出来。
4. ssDNA细胞毒性验证
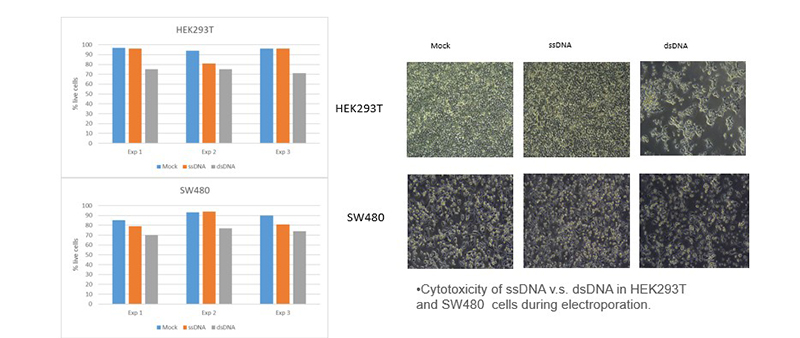
图五:ssDNA和dsDNA细胞毒性验证
每百万细胞电转20pmol的ssDNA和dsDNA,48h后考马斯兰染色检测细胞活力。从结果看ssDNA的细胞毒性明显低于dsDNA。
ssDNA应用案例:
案例分析一:Long ssDNA在小鼠模型构建中的应用

图六:使用Easi-CRISPR将P2A-FlpO融合到Fgf8的3 ‘端[1]
小鼠由于易获得、生长周期短和易操作等优点而被广泛用于人类疾病的建模。研究表明在构建模式小鼠中以Long ssDNA作为“Donor template”时,片段敲入效率达到8.5-100%[1],极大的缩短了建模周期。作者将体外转录合成的crRNA、tracrRNA与Cas9蛋白结合形成RNP复合物和大小为0.8-1.5kb且同源臂长度为60-105nt的ssDNA(P2A-FlpO)同时注射进小鼠受精卵。随后分别对G0代小鼠中Fgf8等6个基因插入位点靶区域进行扩增测序,结果表明Long ssDNA(P2A-FlpO)作为修复模板在小鼠模型中的敲入效率约为25%-67%。
案例分析二: ssDNA 或dsDNA “Donor template”的选择
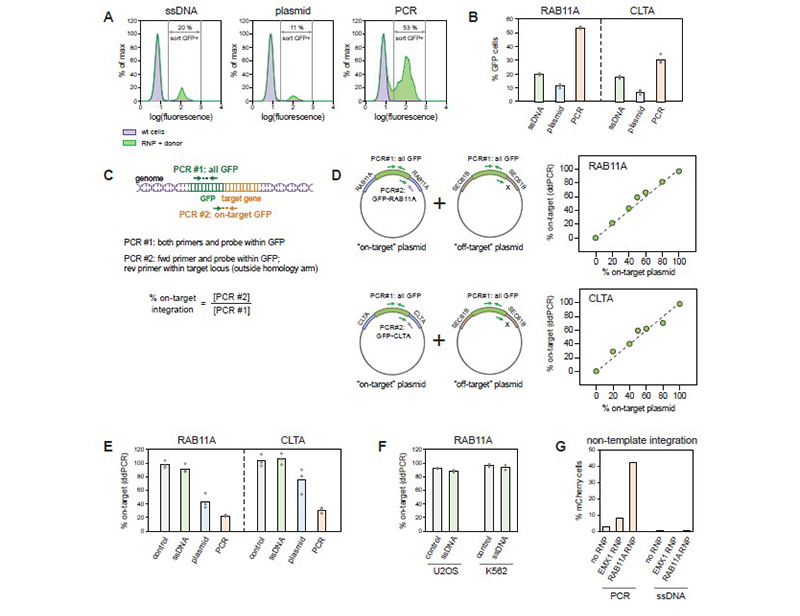
图七: ssDNA、质粒和PCR供体的比较[2]
为了研究不同类型的Donor 片段的敲入效率和特异性,科研人员使用同源臂长度均为150nt且包含GFP序列的ssDNA、质粒和PCR片段靶向HEK293T细胞中的RAB11A 和CLTA位点,通过检测携带GFP的细胞来区分片段敲入效率的高低。研究表明:3种类型的Donor片段敲入效率为PCR片段>ssDNA>质粒;但是进一步的特异性分析表明: ssDNA 表现出更高的特异性,质粒模板较低,PCR产物的特异性最差:在RAB11A 和CLTA位点仅表现出约20-30%特异性,推测可能是由于PCR片段在基因组其他位置出现随机整合。因此, ssDNA无疑是片段敲入实验中最适宜的 “Donor template”。同时作者研究表明在细胞中ssDNA能提高敲入效率到20-40% [2],更易筛选获得片段敲入的突变体。
案例分析三:Long ssDNA同源臂长度和用量对敲入效率的影响
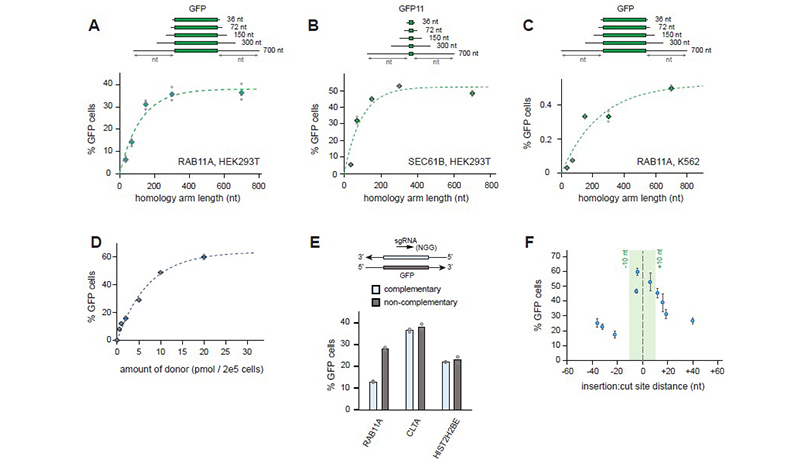
图八: ssDNA供体设计的优化[2]
文章中作者通过使用同源臂长度分别为36nt、72nt、150nt、400nt和700nt的ssDNA在RAB11A和SEC61B中敲入携带GFP的片段来比较不同长度同源臂片段的敲入效率。研究表明:在一定范围内,随着ssDNA同源臂长度的增加敲入效率显著上升。当同源臂长度为300-400nt,修复模板量为20 pmol / 2e5 cells时,其在HEK293T细胞中敲入效率达到最大值的95%,在悬浮细胞株K562中观察到同样的结果。因此,在进行片段敲入实验时,同源臂长度为 400-700 nt、修复模板量为20 pmol / 2e5 cells的ssDNA是好的选择。
参考文献:
[1] Quadros, R.M. et al. Easi-CRISPR: a robust method for one-step generation of mice carrying conditional and insertion alleles using long ssDNA donors and CRISPR ribonucleoproteins. Genome Biol. DOI 10.1186/s13059-017-1220-4.
[2] Li, H,et al. Design and specificity of long ssDNA donors for CRISPR-based knock-in. BioRxiv,Doi https://doi.org/10.1101/178905.
安升达提供的CRISPR/Cas9技术服务的授权来自Broad研究所。
