CRISPR安全评估系列专题(二):临床中的脱靶风险评估
上一次的专题中,我们罗列了学术界几类主流的CRISPR脱靶检测方法,它们原理各不相同,能够找到的脱靶位点也有所区别。在实际应用中,如何选择CRISPR脱靶检测方法,如何选择用于检测的细胞,发现脱靶位点后又如何进行评估,都是需要重点关注的问题。接下来,我们将以几个前沿的CRISPR临床实验为例,阐述一下CRISPR临床实验中的脱靶风险评估策略。
首先,目前进展最快的是诺贝尔奖获得者Emmanuelle Charpentier的公司CRISPR Therapeutics用于治疗β-地中海贫血症和镰刀型贫血症的CTX001,有望在明年提交BLA申请。CTX001是一种体外基因编辑疗法,利用目前已经成熟的方法来获取患者的造血干细胞,在体外实验中进行基因编辑后,再回输到病人体内。所以在评估CTX001的脱靶风险时,研究者选择使用健康志愿者的造血干细胞进行实验,非常好的还原了治疗过程中的真实脱靶情况。而在脱靶检测的实验设计上,CTX001选择了一种预测和再验证的策略。如图1所示,研究者通过gRNA序列进行脱靶预测,找到了171个潜在的脱靶位点;同时采用了细胞内脱靶检测方法GUIDE-seq对健康志愿者的造血干细胞进行检测,发现了52个潜在的脱靶位点。但这两种方法都是对脱靶位点的预测,所以研究者将发现的潜在脱靶位点汇总后,对健康志愿者的造血干细胞进行基因编辑,杂交捕获这些位点并进行高通量测序,测序深度高达15188x。我们一般认为二代测序自身的会有0.1%左右的错误率,所以CTX001的研究者设置了0.2%的检测阈值,检测突变率高于0.2%的位点才认为是真正发生脱靶的位点。在这个标准下,223个潜在的脱靶位点都被认为是安全的,到目前为止,CTX001的临床中没有爆出有与基因编辑直接相关的安全性问题。
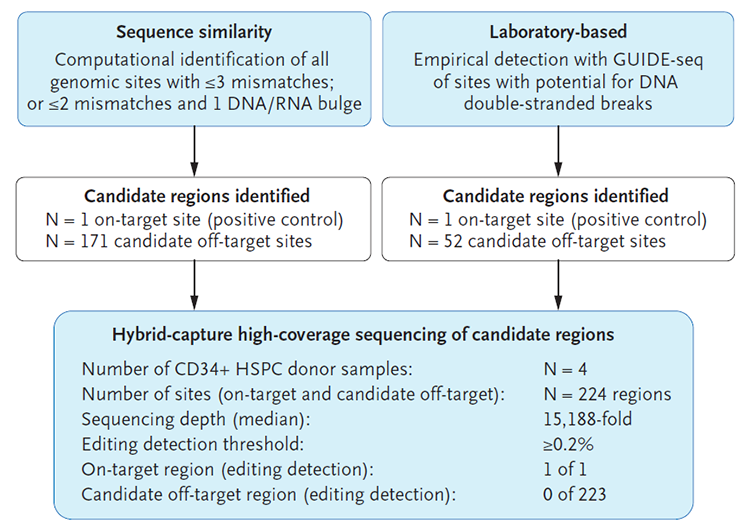
图1 CTX001的脱靶风险评估方案
由于造血干细胞比较容易获取和进行体外实验,所以CTX001的脱靶风险评估可信度较高。但在体内基因编辑疗法中,绝大部分病人的细胞无法直接获取,不仅脱靶风险无法直接评估,甚至治疗后的基因编辑效率也无法检测。所以体内基因编辑疗法需要在临床前实验中进行更为全面的检测,才能保证临床实验的安全和高效。
CRISPR先驱之一,张锋创立的Editas Medicine,其Edit-101是全球首个进入临床的体内基因编辑疗法,用于治疗由CEP290基因内含子突变(c.2991+1655A>G)引起的Leber先天性黑朦10型,通过AAV5的递送方式来靶向视网膜细胞。由于患者的视网膜细胞无法直接获取进行体外实验,研究者设计了更为复杂的脱靶风险评估方案。如图2所示,研究者采用了3种脱靶位点的预测方法,分别是基于gRNA序列的预测、细胞外检测方法Digenome-Seq以及细胞内检测方法GUIDE-Seq。Digenome-Seq对细胞类型没有要求,在增大RNP使用浓度的测试过程中,研究者仅在RNP最高浓度1000nM时找到一个潜在的脱靶位点。在GUIDE-Seq测试中,研究者使用了多种细胞,包括3种永生化细胞系:人骨肉瘤细胞U2OS、人视网膜上皮细胞ARPE-19、人神经母细胞瘤细胞SH-SY5Y,以及2种原代细胞:病人来源的成纤维细胞和T细胞。多种细胞的使用尽可能得弥补了无法在直接用视网膜细胞进行实验的缺陷,按照研究者给出的标准,GUIDE-Seq没有找到一个潜在的脱靶位点。在找到潜在的脱靶位点后,Editas的研究者同样设计了验证实验,除了在U2OS和ARPE-19细胞中转染saCas9质粒进行编辑,他们还获取了非常珍贵的2份死者捐献的视网膜细胞,直接使用EDIT-101的方案进行编辑。借助高通量测序对这些编辑后细胞的潜在脱靶位点进行分析,除了5个NGS测序效果不佳的位点,剩下112个位点的突变率均低于研究者设置的0.1%的最低限度,目前EDIT-101的临床实验也没有爆出有与基因编辑直接相关的安全性问题。
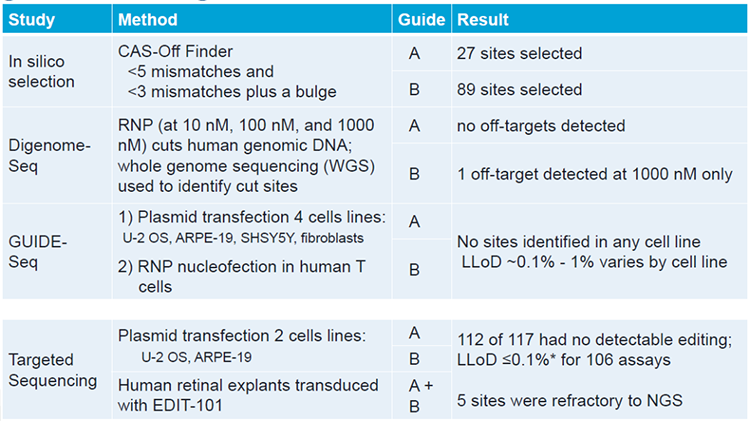
图2 EDIT-101的脱靶风险评估方案
另外一位诺贝尔奖获得者Jennifer A. Doudna创立的Intellia Therapeutics,其临床项目NTLA-2001是目前进展最快的体内基因编辑疗法,用于治疗由TTR基因突变导致的转甲状腺素蛋白淀粉样变性 (transthyretin amyloidosis,ATTR) 疾病,通过LNP递送方式靶向肝脏细胞。如图3所示,在脱靶位点发现阶段,Intellia的研究者采用了3种预测方法,分别是基于gRNA序列的预测、细胞外检测方法SITE-Seq以及细胞内检测方法GUIDE-Seq,总计找到了658个潜在脱靶位点。由于原代肝细胞比较容易获取,研究者设计非常详细的脱靶位点验证实验,以实现肝细胞中靶位点90%编辑效率的LNP用量为1倍剂量,最高测试了27倍剂量的LNP,不同LNP剂量处理肝细胞后都使用高通量测序分析了潜在的658个脱靶位点,最终确定有7个位点是真实的脱靶位点,其中5个位点处在基因间,2个位点位于编码基因的内含子中。这7个脱靶位点突变率和LNP的剂量成正相关,但研究者认为到3倍LNP剂量时,这7个脱靶位点突变率仍低于NGS检测下限,正常治疗过程中脱靶风险不大,到目前为止NTLA-2001的临床中也没有爆出有与基因编辑直接相关的安全性问题。
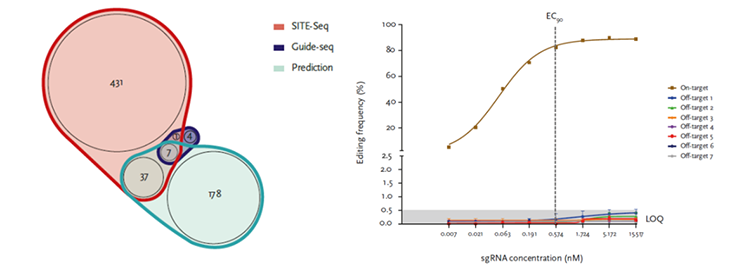
图3 NTLA-2001的脱靶风险评估方案
综合上述3个临床实验的结果,我们可以看出研究者对于CRISPR脱靶风险的高度重视,单一的方法无法准确评估CRISPR的脱靶情况,所以必需结合多种方法预测潜在的脱靶位点,然后再使用高通量测序对这些潜在脱靶位点进行详细分析。3个临床中都采用了细胞内检测方法GUIDE-seq,虽然GUIDE-seq从原理上真实反映了脱靶的发生过程,但是由于其dsODN插入效率的限制,一些低频脱靶位点可能检测不到,最终找到的潜在脱靶位点偏少。从NTLA-2001的实验数据中我们就能看到几种检测方法的差异,基于gRNA序列预测找到了211个潜在脱靶位点,只包括了3个真实的脱靶位点;细胞内检测方法GUIDE-Seq找到了最少的11个潜在脱靶位点,也只包括了3个真实的脱靶位点;而细胞外检测方法SITE-Seq找到了最多的475个潜在脱靶位点,包括了全部7个真实的脱靶位点。从上述实验数据我们不难看出,临床级别的CRISPR脱靶风险评估,最好结合多种检测方法进行。
综合很多进行中的CRISPR基因编辑临床实验,我们能够总结出CRISPR脱靶风险评估的大致思路(图4),可以分为脱靶位点发现、脱靶位点验证和脱靶位点安全性分析这3个阶段。在脱靶位点发现阶段,基于gRNA序列预测和细胞外检测方法可以找到大量脱靶位点,尽量不遗漏任何可能的脱靶位点;而细胞内的检测方法,目前大家普遍选择的GUIDE-seq,虽然能找到的脱靶位点较少,但可信度较高,可以列为重点关注对象。在脱靶位点发现之后,脱靶位点的验证尤为重要,测试条件和测试细胞要尽可能得去还原实际临床中的条件,靶向高通量测序的测序深度也尽可能的高,最终通过统计学的方法评估脱靶位点的真实性。最后,无论哪种CRISPR系统,发生脱靶都是不可避免的事情,少量存在的脱靶位点,还需要进行安全性分析,分析它们在基因组上的位置,与周边基因的距离,是否处在基因组功能元件上。脱靶位点即使处在基因序列上,如果处在内含子序列中,其安全性也需要单独评估,可能对该基因表达不造成影响。
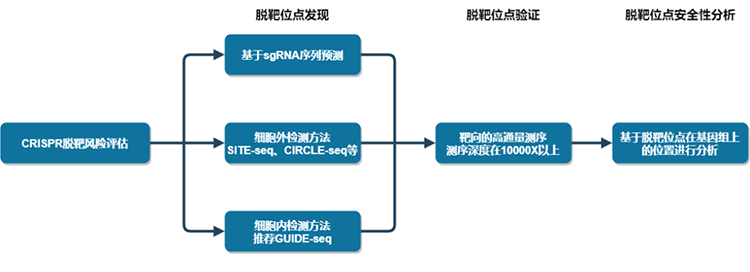
图4 CRISPR脱靶风向评估流程
总结
通过上述过程来评估CRISPR脱靶风险,筛选到脱靶位点较少的gRNA序列,然后再对其脱靶位点进行安全性评估,这样一套严谨的分析方法,可以将临床中的CRISPR脱靶风险降到最低。CRISPR系统最主要的一个变量就是gRNA spacer序列,临床实验中值得投入大量人力物力去筛选获得一条最为合适的gRNA。而gRNA的脱靶风险只是gRNA安全性的一部分考量,即使选择的gRNA没有发生脱靶,gRNA在靶位点引发的随机突变同样可能引发各种风险。如何对gRNA靶位点的突变图谱进行风险评估,是CRISPR安全评估的另一个重要组成部分,我们将在下一篇文章中与大家继续分享这部分的内容。
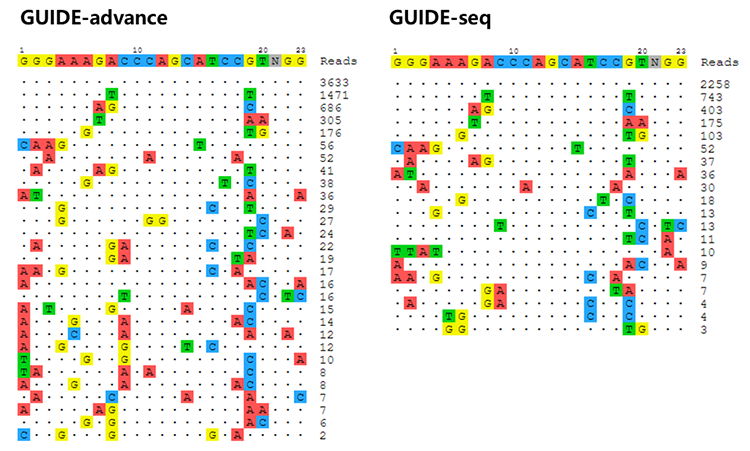
GUIDE-advance细胞内脱靶检测
为帮助科研人员寻找sgRNA的脱靶位点,安升达自主开发了一种新的细胞内脱靶检测方法——GUIDE-advance,改善了传统GUIDE-seq的假阳性、插入效率低等问题,是一种灵敏度高、特异性好,并且可以同时分析多个sgRNA的检测方法。
对GUIDE-advance与GUIDE-seq进行比对,针对同一个spCas9 sgRNA,在相同二代测序数据量的条件下,GUIDE-advance在靶位点和脱靶位点的reads数大约是GUIDE-seq的2倍,并且能够找到更多的脱靶位点。相对于传统方法,GUIDE-advance大大提升了脱靶检测的灵敏度与特异性。
注:本文图片来自参考文献
参考文献
[1] 基因编辑如火如荼!全球首款CRISPR编辑产品即将提交BLA申请,_疗法_治疗_患者 (sohu.com)
[2] Frangoul H, Altshuler D, Cappellini MD, Chen YS, Domm J, Eustace BK, Foell J, de la Fuente J, Grupp S, Handgretinger R, Ho TW, Kattamis A, Kernytsky A, Lekstrom-Himes J, Li AM, Locatelli F, Mapara MY, de Montalembert M, Rondelli D, Sharma A, Sheth S, Soni S, Steinberg MH, Wall D, Yen A, Corbacioglu S. CRISPR-Cas9 Gene Editing for Sickle Cell Disease and β-Thalassemia. N Engl J Med. 2021 Jan 21;384(3):252-260. doi: 10.1056/NEJMoa2031054. Epub 2020 Dec 5. PMID: 33283989.
[3] Maeder ML, Stefanidakis M, Wilson CJ, Baral R, Barrera LA, Bounoutas GS, Bumcrot D, Chao H, Ciulla DM, DaSilva JA, Dass A, Dhanapal V, Fennell TJ, Friedland AE, Giannoukos G, Gloskowski SW, Glucksmann A, Gotta GM, Jayaram H, Haskett SJ, Hopkins B, Horng JE, Joshi S, Marco E, Mepani R, Reyon D, Ta T, Tabbaa DG, Samuelsson SJ, Shen S, Skor MN, Stetkiewicz P, Wang T, Yudkoff C, Myer VE, Albright CF, Jiang H. Development of a gene-editing approach to restore vision loss in Leber congenital amaurosis type 10. Nat Med. 2019 Feb;25(2):229-233. doi: 10.1038/s41591-018-0327-9. Epub 2019 Jan 21. PMID: 30664785.
[4] PowerPoint Presentation (editasmedicine.com)
[5] Gillmore JD, Gane E, Taubel J, Kao J, Fontana M, Maitland ML, Seitzer J, O’Connell D, Walsh KR, Wood K, Phillips J, Xu Y, Amaral A, Boyd AP, Cehelsky JE, McKee MD, Schiermeier A, Harari O, Murphy A, Kyratsous CA, Zambrowicz B, Soltys R, Gutstein DE, Leonard J, Sepp-Lorenzino L, Lebwohl D. CRISPR-Cas9 In Vivo Gene Editing for Transthyretin Amyloidosis. N Engl J Med. 2021 Aug 5;385(6):493-502. doi: 10.1056/NEJMoa2107454. Epub 2021 Jun 26. PMID: 34215024.
