CRISPR安全评估系列专题(一):脱靶位点检测大盘点
近几年,细胞和基因治疗(cell & gene therapy,CGT)领域发展迅猛,在多个方向取得了重大突破,以anti-CD19和BCMA为代表的多种CAR-T免疫细胞疗法攻克了一部分血液瘤,多种AAV疗法也给一些难以成药的罕见病提供了有效的治疗方案。另一方面,基于CRISPR基因编辑技术的众多基因疗法也进展迅速,首个体外基因编辑疗法最快今年年底能够上市(CRISPR Therapeutics CTX001)[1],体内基因编辑疗法取得了不错的临床数据(Intellia Therapeutics NTLA-2001)[2],使用CRISPR技术制造的通用型CAR-T疗法也是免疫细胞疗法发展的一大趋势(Caribou Biosciences CB-010)[3]。
CRISPR基因编辑治疗发展如火如荼,但也有不少人对基因治疗的安全性提出担忧,由于CRISPR技术是直接改变基因组DNA,其中任何的改变都会被永久保留下来,所以CRISPR对于DNA序列的任何改变都需要严谨的安全评估。根据目前已经公开的FDA关于基因治疗的指导文件[4],对于基因治疗安全性的评估可以简单总结为两个方面:
- Assessment of genomic integrity, including chromosomal rearrangements, large insertions or deletions, integration of exogenous DNA, and potential oncogenicity or insertional mutagenesis.
- Identification of off-target editing activity, including the type, frequency, and location of all off-target editing events.
由于目前大部分CRISPR基因编辑治疗方案是基于CRISPR引起的DNA双链断裂和随机突变进行基因敲除,所以第一类风险主要是指靶位点的随机突变引发的安全风险,如果出现大片段丢失、染色体重排等异常变化,都存在巨大的安全隐患。另一方面,CRISPR基于sgRNA与基因组序列互补定位到靶位点,不论哪种CRISPR系统,都存在sgRNA序列不完全匹配但能够结合基因组的脱靶现象,CRISPR定位到脱靶位点引发序列改变就属于第二类风险。CRISPR技术诞生刚过10年,期间迅速取代TALEN和ZFN,成为学术界通用的基因编辑技术,也彻底改变了我们的生物学研究方法。为提高CRISPR技术的安全性,特别是往临床应用发展时的安全性问题,研究者们一直以提高靶位点的编辑效率和准确性,同时降低脱靶位点编辑发生概率为目标,来优化CRISPR技术。另一方面,研究者们也开发了大量检测方法,用于检测CRISPR的靶向风险和脱靶风险,用于早期sgRNA序列的筛选,以期望获得一个最为安全的sgRNA。
脱靶位点检测技术盘点
围绕大家关心的CRISPR基因编辑安全性问题,我们首先对CRISPR脱靶位点检测技术进行一次大盘点,在sgRNA设计阶段需要如何做才能够尽可能地避免脱靶现象的发生。最简单的脱靶位点检测方法是全基因组测序(WGS)[5],但在实际使用中,即使测序深度达到100X,也很难发现一些低频率的脱靶位点,同时测序成本却非常高。为弥补WGS的这些缺陷,常见的脱靶位点检测技术都需要对脱靶位点进行捕获富集,来提高检测灵敏度,降低测序成本。按照实验原理的不同,脱靶位点检测技术可以分为细胞外、细胞内和其他特殊方法这3类。
- 细胞外检测方法
细胞外检测方法最为直接,将基因组提纯后进行CRISPR体外切割实验,再捕获发生脱靶切割的位点即可。
1) Digenome-seqDigenome-seq[6]是较早提出的一类细胞外脱靶位点检测方法(图1左),提纯的基因组DNA被Cas9/sgRNA切割后,再经过标准的全基因组测序流程获得测序数据,之后需要较为复杂的生物信息学分析才能够获得CRISPR切割位点的信息。总体来讲,这种方法测序成本较高,并且检测灵敏度也有限。
2) SITE-seq为了有效检测到低频脱靶位点,一般需要在建库时定向捕获CRISPR切割位点。SITE-seq[7]就是这样一种测序方法(图1右),提纯的基因组DNA经过Cas9/sgRNA切割后,在切割位点末端连接带Biotin的接头;再随机打断基因组,在另一端连接第二个接头;最后经过链霉亲和素磁珠纯化,只有第一轮被Cas9切割的DNA才能够被纯化并测序,实现了CRISPR切割位点的富集。该方法虽然提高了脱靶位点的检测灵敏度,但有着较高的实验要求,每次需要投入大量的基因组DNA,并且无法区分提纯基因组DNA时发生的随机断裂和Cas9的切割,导致产出较为明显的背景信号。同时,由于第一轮Biotin接头只会接在Cas9切割位点的一侧,导致测序结果里只能看到脱靶位点一侧的序列。
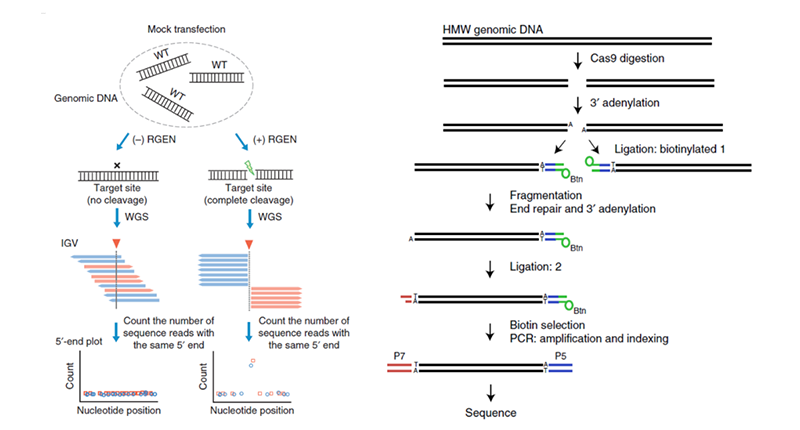
图1 Digenome-seq(左)和SITE-seq(右)原理图
3) CIRCLE-seq和CHANGE-seq与SITE-seq同期发表的CIRCLE-seq[8]一定程度上解决了SITE-seq的一些问题。CIRCLE-seq(图2)的第一步是将纯化后的基因组DNA随机打断成300bp左右的片段,然后首尾相连成环状DNA,这种方法很好地避免了DNA随机断裂造成的背景噪声。实验的第二步是用Cas9切割环状DNA,再连上接头并进行双端测序,这样就可以在一次测序中同时获取切割位点的两侧序列,弥补了SITE-seq的另一个缺点。CIRCLE-seq从总体上来说要优于SITE-seq,但是将基因组DNA随机打断后成环的效率并不高,所以同一作者在3年后发表了CHANGE-seq[9],用Tn5一步法打断基因组并加接头,提高了成环效率,降低了起始基因组DNA的用量。
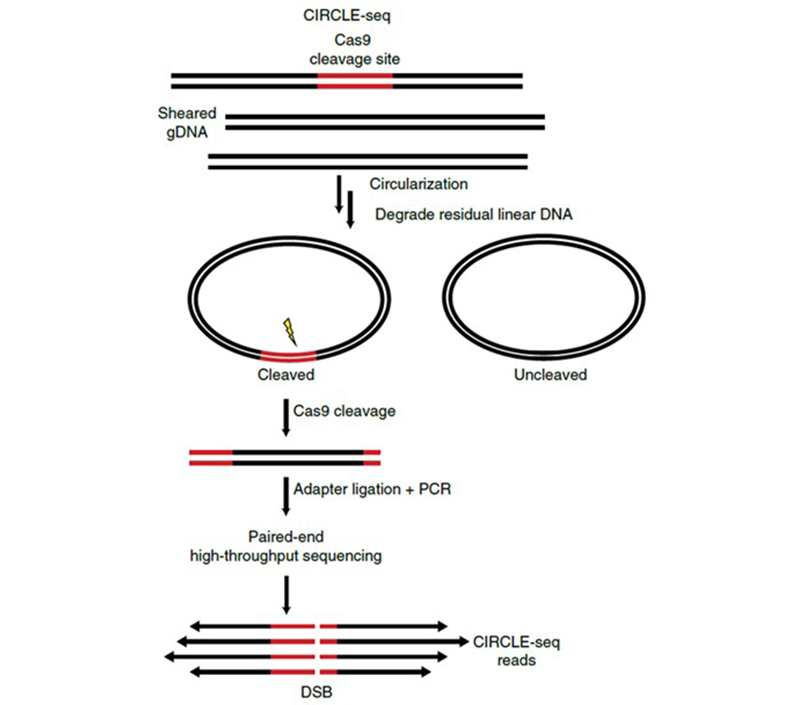
图2 CIRCLE-seq原理图
2. 细胞内检测方法由于提纯的基因组已经消化去除了组蛋白,结构较细胞内的染色质更为松散,理论上容易找到更多的脱靶位点。为捕获CRISPR在细胞内工作时真实发生的脱靶切割,研究者们也设计了一些细胞内的脱靶位点检测方法。
1) Guide-seqCRISPR造成DNA双链断裂后,由于DNA修复机制的存在,断裂处很快就会重新连接,这时候提纯基因组DNA很难保留CRISPR造成的DNA断裂位点。所以为了记录细胞内真实的CRISPR脱靶切割,研究者们设计了Guide-seq[10],利用NHEJ的DNA修复机制,将一段双链短核苷酸序列(dsODN)连接到CRISPR造成的DNA双链断裂处,相当于连接了第一轮接头,然后再正常打断基因组,在另一侧连接第二轮接头。通过这种方式构建的文库,就可以获取脱靶位点一侧的序列(图3)。虽然每次CRIPSR导致的DNA双链断裂并不一定都会连接dsODN,但反过来说,越容易连接dsODN的位点,其发生脱靶切割的概率越高。通过Guide-seq找到的脱靶位点偏少,但一般都是可信度较高的脱靶位点,Guide-seq也是目前比较公认的一种脱靶检测方法。
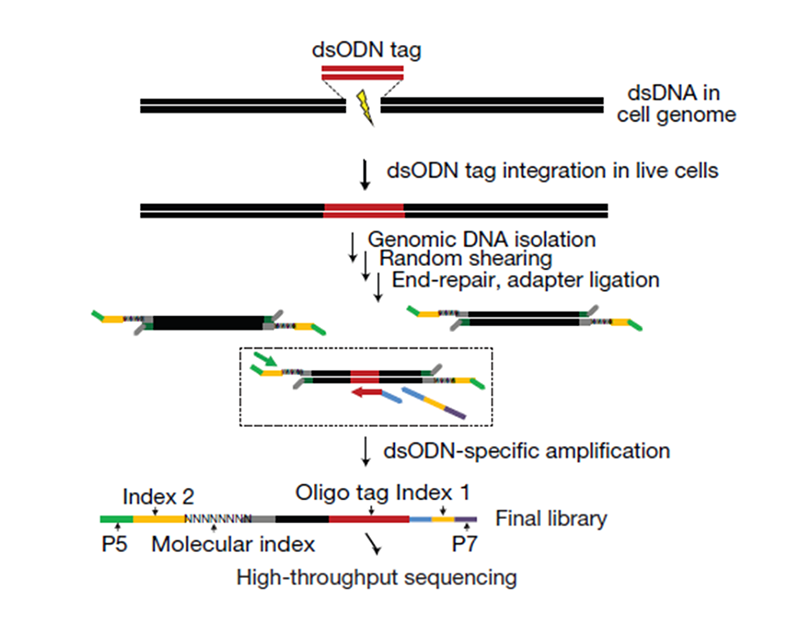
图3 Guide-seq原理图
2) Discover-seq细胞内的脱靶切割一般无法直接捕获,除了Guide-seq这种连接dsODN来间接记录脱靶位点的方法外,研究者们还想出一种蛋白介导的方法Discover-seq[11]。由于DNA双链断裂后细胞会启动DNA修复机制,大量蛋白参与到断裂处的修复,所以研究者就对相关蛋白进行筛选,找到其中MRE11结合DNA的位点与DNA双链断裂的相关性最好。DISCOVER-seq便是通过MRE11的CHIP-seq(染色质免疫共沉淀)来反映CRISPR的脱靶位点。但是由于CHIP-seq实验本身的难度较高,DISCOVER-seq这类方法的接受度并不高。
3. 其他特殊方法CRISPR技术经过这么多年的发展,其应用已经不局限于造成DNA双链断裂,一些不要切割DNA双链的CRISPR衍生技术(如碱基编辑、先导编辑和CRISPRi/a),可以在不引发随机突变的情况下,对基因组进行精准编辑或者调控。这些技术所使用nCas9或dCas9仅结合或者切割双链中的一条链,之前的脱靶检测方法都不能直接适用。这些CRISPR衍生技术中,CRISPR产生的脱靶可以被细分为两个部分,其一是Cas9/sgRNA结合DNA导致的脱靶,这部分信息使用同样序列的sgRNA进行野生型Cas9脱靶位点检测能够间接得到;第二种脱靶来自于这些技术所携带的功能基团,如碱基编辑的脱氨酶和先导编辑的逆转录酶,不依赖sgRNA结合基因组DNA的情况下产生的脱靶。为检测第二类脱靶现象,研究者需要针对每种技术设计专门的检测方法。
1) R-loop seq在碱基编辑开发早期,虽然靶位点的编辑效率很高,但研究者也很快发现脱氨酶会在游离的时候随机修饰暴露的DNA单链,导致大量的脱靶位点。为降低这类脱靶现象的发生,研究者设计了R-loop seq[12],以dSaCas9结合DNA形成R-loop,暴露出DNA单链,为碱基编辑的脱氨酶提供一个固定的脱靶位点,然后以该位点的编辑效率反映碱基编辑脱氨酶的脱靶程度(图4左)。R-loop seq虽然不是一种真正意义上的脱靶位点检测方法,但能为碱基编辑脱氨酶的脱靶提供一种度量方法,以便于之后的脱氨酶点突变优化,来降低脱靶的发生几率。
2) Detect-seqCRISPR衍生技术由于其复杂性,检测其脱靶位点的核心思路是捕获实验过程中的关键中间产物或者终产物。这种设计思路下,目前较为成功的是检测碱基编辑CBE脱靶位点的Detect-seq[13]。CBE的原理是将dC脱氨变为dU,迫使其对侧的dG变为dA,最终将碱基对从C-G转变为T-A。Detect-seq便是针对中间态的dU,使用Uracil DNA Glycosylase (UDG),去除U碱基后,替换为带Biotin的U碱基,捕获碱基编辑的脱靶位点(图4右),并且sgRNA依赖和sgRNA不依赖的脱靶位点都能找到。该方法类似检测DNA甲基化的重亚硫酸盐测序法,通过处理特定的碱基,可以捕获全基因组上的CBE脱靶位点。
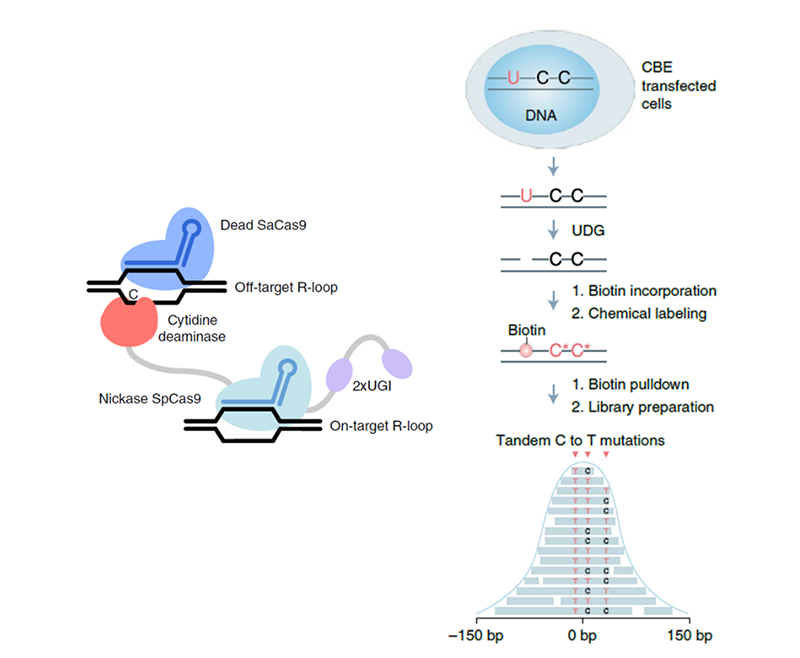
图4 R-loop seq(左)和Detect-seq(右)原理图
总结
不论在科研还是临床中,CRISPR技术的使用都带来了非常高的回报,也同时伴随着各种风险,这其中脱靶现象就研究者们最为担心的一类风险。本文中我们盘点了多种主流的CRISPR脱靶位点检测方法,但没有一种方法是适用于所有CRISPR技术的脱靶检测,一个项目中只使用一种脱靶检测方法也是不足够的。如何在实验中,特别是临床试验中全面评估CRISPR的脱靶风险,需要将多种脱靶检测方法有效组合。同时,每一条sgRNA都不可避免地存在脱靶位点,如何评估这种风险,如何降低这种风险,具体的解决方案我们会在下一期的文章中,用临床实例来为大家介绍。
注:本文图片均来源于参考文献参考文献
[1] 基因编辑如火如荼!全球首款CRISPR编辑产品即将提交BLA申请,_疗法_治疗_患者 (sohu.com)
[2] Intellia和再生元公布NTLA-2001最新1期临床试验数据_新浪医药新闻 (sina.com)
[3] 速递|开创多个第一!即用型CAR-T疗法达到100%总缓解率_腾讯新闻 (qq.com)
[4] Human Gene Therapy Products Incorporating Human Genome Editing | FDA
[5] Veres A, Gosis BS, Ding Q, Collins R, Ragavendran A, Brand H, Erdin S, Cowan CA, Talkowski ME, Musunuru K. Low incidence of off-target mutations in individual CRISPR-Cas9 and TALEN targeted human stem cell clones detected by whole-genome sequencing. Cell Stem Cell. 2014 Jul 3;15(1):27-30.
[6] Kim D, Bae S, Park J, Kim E, Kim S, Yu HR, Hwang J, Kim JI, Kim JS. Digenome-seq: genome-wide profiling of CRISPR-Cas9 off-target effects in human cells. Nat Methods. 2015 Mar;12(3):237-43, 1 p following 243.
[7] Cameron P, Fuller CK, Donohoue PD, Jones BN, Thompson MS, Carter MM, Gradia S, Vidal B, Garner E, Slorach EM, Lau E, Banh LM, Lied AM, Edwards LS, Settle AH, Capurso D, Llaca V, Deschamps S, Cigan M, Young JK, May AP. Mapping the genomic landscape of CRISPR-Cas9 cleavage. Nat Methods. 2017 Jun;14(6):600-606.
[8] Tsai SQ, Nguyen NT, Malagon-Lopez J, Topkar VV, Aryee MJ, Joung JK. CIRCLE-seq: a highly sensitive in vitro screen for genome-wide CRISPR-Cas9 nuclease off-targets. Nat Methods. 2017 Jun;14(6):607-614.
[9] Lazzarotto CR, Malinin NL, Li Y, Zhang R, Yang Y, Lee G, Cowley E, He Y, Lan X, Jividen K, Katta V, Kolmakova NG, Petersen CT, Qi Q, Strelcov E, Maragh S, Krenciute G, Ma J, Cheng Y, Tsai SQ. CHANGE-seq reveals genetic and epigenetic effects on CRISPR-Cas9 genome-wide activity. Nat Biotechnol. 2020 Nov;38(11):1317-1327.
[10] Tsai SQ, Zheng Z, Nguyen NT, Liebers M, Topkar VV, Thapar V, Wyvekens N, Khayter C, Iafrate AJ, Le LP, Aryee MJ, Joung JK. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nat Biotechnol. 2015 Feb;33(2):187-197.
[11] Wienert B, Wyman SK, Richardson CD, Yeh CD, Akcakaya P, Porritt MJ, Morlock M, Vu JT, Kazane KR, Watry HL, Judge LM, Conklin BR, Maresca M, Corn JE. Unbiased detection of CRISPR off-targets in vivo using DISCOVER-Seq. Science. 2019 Apr 19;364(6437):286-289.
[12] Doman JL, Raguram A, Newby GA, Liu DR. Evaluation and minimization of Cas9-independent off-target DNA editing by cytosine base editors. Nat Biotechnol. 2020 May;38(5):620-628.
[13] Lei Z, Meng H, Lv Z, Liu M, Zhao H, Wu H, Zhang X, Liu L, Zhuang Y, Yin K, Yan Y, Yi C. Detect-seq reveals out-of-protospacer editing and target-strand editing by cytosine base editors. Nat Methods. 2021 Jun;18(6):643-651.
