基因编辑的极限在哪里——迷你核酸酶IscB和TnpB的发现
CRISPR/Cas9技术自2012年发表以来,基因编辑技术获得了飞速发展。相比于上一代的基因编辑技术ZFN和TALEN,CRISPR/Cas9技术最大的优势是由RNA引导进行核酸内切。相比于ZFN和TALEN需要进行复杂的蛋白序列重编程,CRISPR/Cas9只需要进行sgRNA的重编程,就可以完成对不同DNA序列的识别和切割。此后发现的分子量更小的CRISPR/Cas12系统和识别RNA的CRISPR/Cas13系统,进一步拓宽了CRISPR技术的应用范围。
纵观CRISPR近10年的发展历程,目前被科研界广泛接受和应用的仍然是最早发表的spCas9(Streptococcus pyogenes Cas9)。spCas9有很多优点:
适合在哺乳动物,特别是人的细胞内工作,后续发现的Cas12等蛋白基因编辑效率并没有优于spCas9;
PAM序列相对简单,NGG序列可以粗略认为出现几率是1/8,同时后续的蛋白工程改造进一步拓展了spCas9的PAM序列,比如spCas9-NG只需要识别一个G碱基[1],而SpRYCas9已经没有了PAM序列的要求[2];
Cas9具有RuvC和HNH两个DNA酶活性位点,分别负责一条DNA单链的切割,突变其中任何一个位点,都可以将Cas9变成只切单链的Cas9 nickase,碱基编辑和先导编辑等Cas9重要新技术都是基于Cas9 nickase设计[3][4][5];而Cas12蛋白仅有一个RuvC酶活性位点,负责DNA双链的切割,也就不存在Cas12 nickase这种形式;
spCas9的sgRNA也是目前改造最多的sgRNA序列,可以在多个位置添加新的功能序列,而不影响sgRNA的基本功能[5][6]。
但spCas9也存在一个致命的缺点,其CDS序列长度超过4.1kb,再算上启动子和polyA序列,它很难通过腺相关病毒AAV(容量上限在4.7kb左右)来递送。即使用慢病毒(容量上限在8kb左右)来递送,也会占用一大部分容量,压缩后续的改造空间,这个问题制约了spCas9在一部分临床实验上的发展。但其他一些核酸酶如saCas9和Cas12,除了更小之外,综合能力仍然不如spCas9。所以目前绝大部分的基因编辑科研和临床实验仍然在使用spCas9,但生物学界也在期盼着一个更小的核酸酶能够完美替代spCas9。
今年9月先后在Science和Nature发表的两篇文章[7][8],就向我们揭示了一类新的基因编辑系统,来自于IS200/605转座子家族的IscB和TnpB,它们在微生物中广泛分布,被认为是CRISPR/Cas9和Cas12系统的起源。它们的最大特点是非常短的CDS序列,在1.2kb左右,体积不到spCas9的1/3,留下了巨大的改造空间。

这里重点解读一下Zhang Feng课题组发表的Science文章,其研究的IscB蛋白被认为是CRISPR/Cas9系统的起源,具备很多类似的特性。从蛋白结构预测分析中,我们能够看到IscB同样存在RuvC和HNH两个酶活性位点(图1左);借助RNA测序和RNA-pulldown等技术手段,研究者也找到了IscB对应的sgRNA,其结构比spCas9的sgRNA还要更加复杂,被作者称为ωRNA(图1右)。
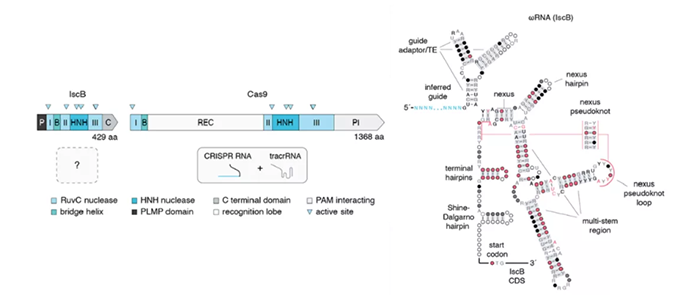
图1IscB和ωRNA的结构预测
在确定了IscB和ωRNA的结构之后,研究者就自然而然地想分析其DNA酶切活性。通过体外DNA切割实验,研究者发现IscB具备由ωRNA引导识别(图2左)并切割特定DNA双链序列的能力,其PAM序列也被作者称为TAM(target-adjacent motif)。不同于spCas9的是,IscB的切口是粘性末端(图2右)。

图2IscB切割DNA双链的模式图
由于IscB具有RuvC和HNH两个酶活性位点,研究者也通过点突变实验验证了IscB的nickase能力,突变其中任何一个酶活性位点都能产生相应的DNA单链切割能力(图3)。

图3IscB具备nickase能力
衡量核酸酶作为基因编辑工具最重要的指标是其在真核细胞内切割DNA的能力,所以研究者在文章的最后也分析了多个物种来源的IscB蛋白编辑人细胞基因组序列的能力。从目前的结果来看,部分IscB蛋白能够编辑人细胞的基因组序列,但编辑效率还明显低于spCas9,最高也只有2%左右(图4)。

图4IscB具备编辑人细胞基因组的能力
综合来看,对于IscB的研究加深了我们对于CRISPR/Cas9系统的起源进化的认识,同时也为我们提供了一类全新的基因编辑工具酶。IscB已经具备了很多spCas9的优点,比如有两个酶活性位点,分别负责一条DNA单链的切割;又比如ωRNA的结构较为复杂,便于进行改造。而IscB最大的优势还是其非常小的体积,为递送提供了极大的便利,同时也就腾出了巨大的改造空间。IscB在微生物中分别广泛,相信在不久的将来,应该能够筛选到在真核细胞中编辑效率和靶向特异性达到spCas9水平的IscB,进一步加速基因编辑科研和临床的发展。
参考文献
[1] EngineeredCRISPR-Cas9 nuclease with expanded targeting space. Science. 2018 Sep21;361(6408):1259-1262. doi: 10.1126/science.aas9129.
[2] Unconstrainedgenome targeting with near-PAMless engineered CRISPR-Cas9 variants.Science. 2020 Apr 17;368(6488):290-296. doi: 10.1126/science.aba8853.
[3] Programmableediting of a target base in genomic DNA without double-stranded DNAcleavage. Nature. 2016 May 19;533(7603):420-4. doi:10.1038/nature17946.
[4] Programmablebase editing of A•T to G•C in genomic DNA without DNA cleavage.Nature. 2017 Nov 23;551(7681):464-471. doi: 10.1038/nature24644.
[5]Search-and-replace genome editing without double-strand breaks ordonor DNA. Nature. 2019 Dec;576(7785):149-157. doi:10.1038/s41586-019-1711-4.
[6] Genome-scaletranscriptional activation by an engineered CRISPR-Cas9 complex.Nature. 2015 Jan 29;517(7536):583-8. doi: 10.1038/nature14136.
[7] The widespreadIS200/605 transposon family encodes diverse programmable RNA-guidedendonucleases. Science, 2021, doi:10.1126/science.abj6856.
[8]Transposon-associated TnpB is a programmable RNA-guided DNAendonuclease. Nature. 2021 Oct 7. doi: 10.1038/s41586-021-04058-1.
