浅谈合成生物学之超大基因如何高效组装
21世纪,合成生物学领域的研究如火如荼,已然成为最受关注的研究主题之一。那么,什么是合成生物学?
合成生物学是指生物学家和工程师,通过重新设计和改造现有的生物系统,达到我们一些特定的应用需求(Fig. 1)。比如,通过改造微生物,生产绿色能源燃料或生产高产量、生产便利、价格便宜的生物药。

Fig.1Illustration of synthetic biology.
早在几千年前,我们就能通过微生物发酵来生产如啤酒,酸奶等各种产品。但传统的生产工艺存在很多的局限性,例如:副产物带有毒性,成本高,产量低,培养时间长等。随着DNA合成技术和实验室自动化的不断创新,合成生物学能够重新设计和构建自然生物系统,使我们有能力去生产一些有特殊要求的产品。更多的投资者也参与到了这一领域,他们相信,这些理想化的生物系统和新颖的生物细胞生产工艺可以在食品、燃料、医药、蛋白质和材料等领域中代替很多原来的化学和工业流程(Fig.2)。例如:1LanzaTechR使微生物能够将废气(例如二氧化碳或甲烷)转化为燃料和化学品。
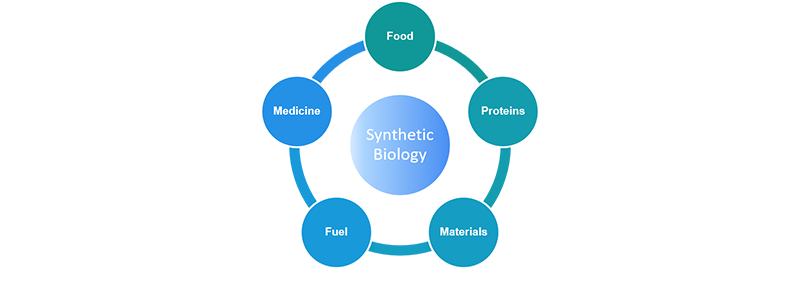
Fig.2 New cell-produced products by means of synthetic biology.
“设计-构建-测试-学习-再构建”的研究循环是合成生物学中基因组设计和合成的一般过程(图3)。2首先,使用计算机软件辅助设计要合成的DNA构建。设计的DNA分为1–1.5kb的可合成片段(synthons)。合成子可以通过化学合成的带有overlap的单链寡核苷酸进行组装拼接合成。多个合成子也可以继续组装成更大的DNA元件。组装好的DNA需要进行序列验证,并将其转化到细胞进行功能验证。根据验证结果,进行修改,重复测试循环,直到获得所需要功能的DNA构建体。这种设计、构建、测试、学习以及修改/重复过程是合成生物学的基础。为了减少时间,减少实验室工作和降低成本,需要相应的自动化以简化总流程。
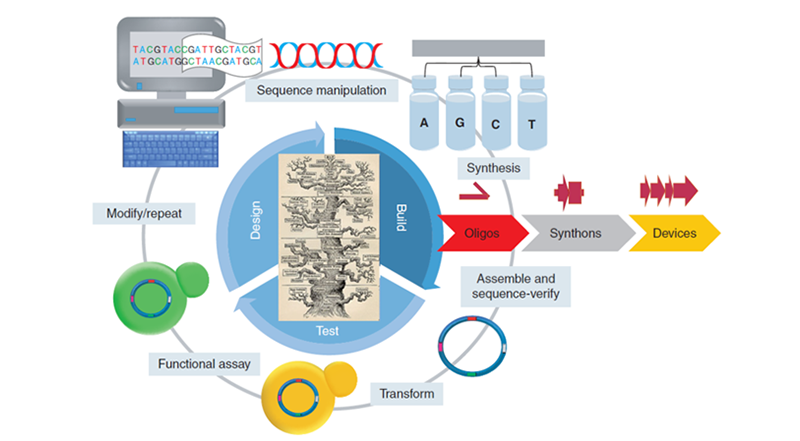
Fig.3 The synthetic biology test cycle.2
2000-2010年,学术界发表了大量关于合成生物学的研究论文,其中有很多天马行空富有远见的思考让人印象深刻。在他们看来,研究人员可以组装创造一个全新的生命体,并赋予它特殊的功能性。然而,生命元件的组装不像电路组装一样简单。2010年,Nature发表一篇报道“Five Hard Truths for Synthetic Biology”,它指出生物工程技术的缓慢进展正延缓合成生物学的发展(图4)3。虽然我们知道,生命体中的任何元件都是由DNA翻译而来,但很多元件的特性我们了解的并不透彻。这些元件的功能也可能会因为时间,位置,条件等的不同而改变。即使我们知道每一种元件的的功能,但我们将这些不同的生物元件组装起来以后,它们的功能也可能会丧失或者互不兼容。2006年,Christopher Voigt组装了一些主要来自枯草芽孢杆菌的遗传物质,并将它们转移到大肠杆菌中,希望在人工控制的条件下对这些基因功能进行分析研究,但并没有得到预期的结果。此外,随着构建变得越来越大,组装和验证过程也变得越来越困难。花费在上面的时间和金钱也是巨大的。例如:Keasling的团队开发了抗疟药物前体青蒿素酸在酵母中的生产流程,包括12个相关基因的发现、功能研究、调控等。据估计,这个项目开发完成耗费了相当于150个人一年的工作量。

Fig.4 Five Hard Truths for Synthetic Biology.3
2010-2020年,随着生物技术和生物工程在合成生物学方面的发展,合成生物学界终于不再只是那些天马行空的想象,而是取得了一些实实在在的成就。2010年是合成生物学界应该被铭记的一年,这一年J.Craig Venter Institute (JCVI)的一个团队首次人工合成了完整的支原体基因组(Fig.5)。支原体是已知最小的生物体,很适用于做基因组研究。这个首次被合成并且成功组装的超大DNA是丝状支原体基因组,它的全长有1.08M,被称为JCVI-syn1.0。将这个人工合成的基因组移植到近缘物种山羊支原体受体中后,新的细胞表现出了预期表型特征,而且只受移植染色体的控制7。从头合成的基因组具有天然的生物特征,这对合成生物学来说是一个重大的突破。
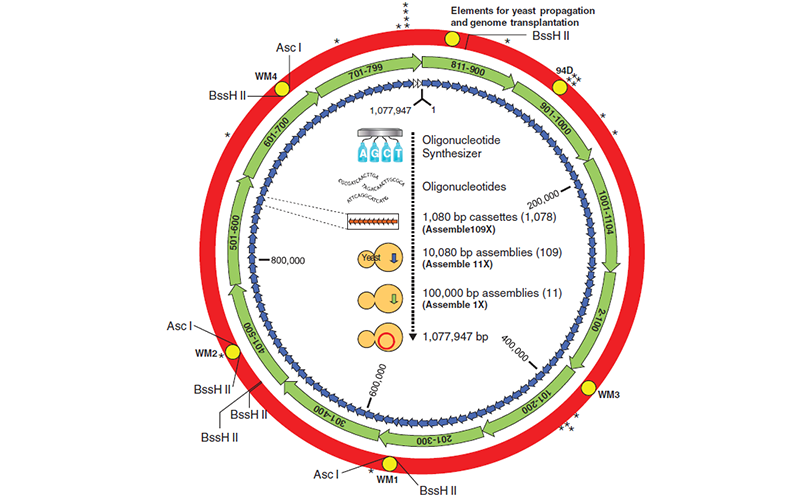
Fig.5 The assembly of a synthetic M.mycoidesgenome in yeast.7
自从JCVI-syn1.0成功合成以来,JCVI研究团队一直在努力尝试将这个基因组简化。他们将JCVI-syn1.0基因组分成8个片段,每个片段都在其余7个片段保持syn1.0基因组环境不变的情况下的下单独进行活性测试,结合优化后的T5转座子突变方法,终于在2016年,成功合成了JCVI-syn3.0(Fig.6)8。JCVI-syn3.0是经过对JCVI-syn1.0整体的重新优化设计合成后,能实现细胞活性的最小的基因组(全长531kb,包含473个基因)。JCVI-syn3.0细胞的基因组比自然界中发现的任何自主复制的细胞都要小,只保留了生存必要的基因,是进行基因组设计和功能性研究理想化的平台。8
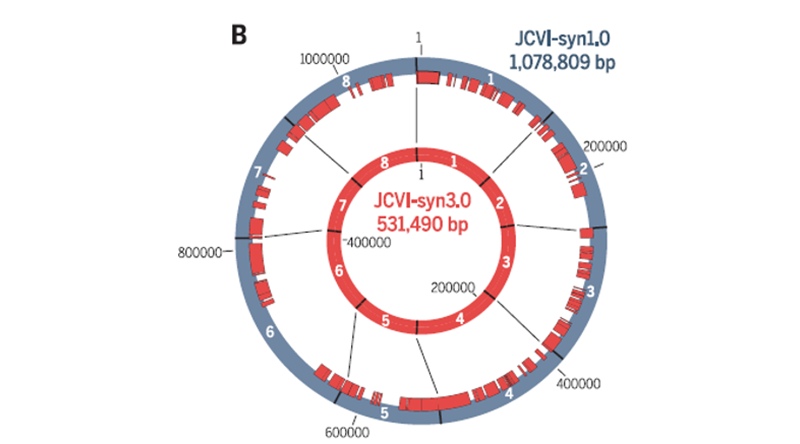
Fig.6 Comparison of JCVI-syn1.0 (outer blue circle) with JCVI-syn3.0(inner red circle), showing the division of each into eight segments.The red bars inside the outer circle indicate regions that areretained in JCVI-syn3.0.8
2007年,Jef D.Boeke教授发起了一个全球化的真核生物研究合作项目-人工合成酵母基因组计划Sc2.0。该项目分布在全球许多国家,其中美国和中国分别占总合成的28%和39%。Sc2.0旨在对酿酒酵母基因组的16条染色体进行从头人工化学合成,从而为系统性研究真核生物染色体提供应用平台9。在Sc2.0的设计过程中,虽然对基因序列进行了碱基删除、插入和替换,但原则上要保持合成菌株与天然菌株的相同表型。同时,也要保证基因组的稳定性,例如:去除了重复序列,优化tRNA和内含子序列,用UTC替换掉酵母染色体的天然端粒,引入和移除部分限制性酶切位点,使GC含量正常化等9。根据具体的需求,对野生型的基因组序列进行了优化,来增强遗传灵活性。例如:将TAG替换为TAA,引入loxPsym位点构建诱导进化系统,引入PCRTag水印筛选标签等9。
截至2017年,Sc2.0的团队已经合成和组装了超过1/3(6.5条染色体)的酵母基因组9。其中,天津大学元英进教授的团队几乎同时完成了V号和X染色体的合成。10,11作为一家生命科学领域的供应商,GENEWIZ在该项目完成的过程中,提供了如:Oligos、DNA片段(~5kb)、Sanger测序等一系列科研服务。
化学合成如基因组这样的DNA大构建,对合成生物学具有重要的意义。然而,一般的科研机构都只能从供应商那里订购到引物或DNA片段,然后自己进行拼接。GENEWIZ作为全球生命科学领域最可靠的供应商之一,在构建大分子DNA方面有着丰富的经验。GENEWIZ在全球范围内首次合成了如人线粒体基因组(约16.5kb)和牛磺酸牡蛎叶绿体基因组(约72kb)等。基因组的合成主要依赖于DNA由小到大的高效组装,以150kb构建为例。构建流程如下Fig.7,是由小到大,分步,依次组装的。
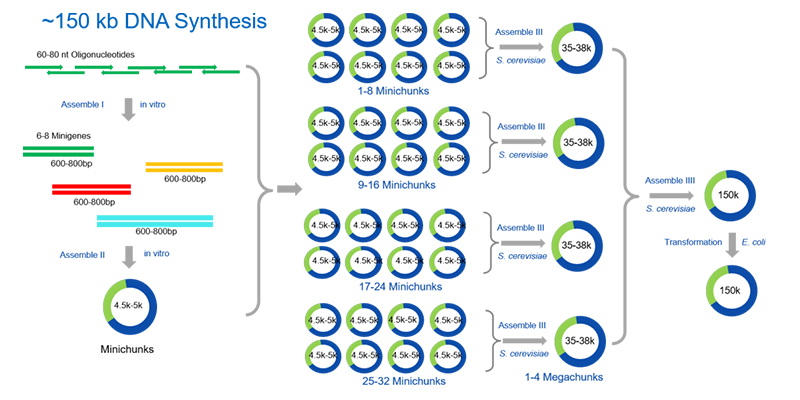
Fig.7 Assembly flow of large DNA constructs (~150 kb) in GENEWIZ.
第一步的组装,是通过两轮的多重重叠延伸PCR,将多条前后有碱基互补配对重叠区的Oligos(60~80 nt)拼接起来,得到约(600~800bp)的Mini geneDNA片段。
第二步的组装,是将这些(600~800bp)的MinigeneDNA片段拼接成Mini chunks(4.5~5 kb)的片段。近年来除了传统的组装方案,还出现了几种新的DNA组装方法。如:BioBrick组装、GoldenGate组装、Gibson组装、TA克隆、TOPO-TA克隆等。在实际应用中,可以根据DNA的长度,限制性酶切位点,重复序列和GC含量的不同,采用一种或几种方案结合的组装方法。
在得到这些Minichunks(4.5~5 kb)的片段后,再利用体内同源重组一步步拼接成更大的chunksDNA片段(35~38kb),甚至可以一步直接拼成最终的构建(~150kb)。体内同源重组最早是用于两条DNA链进行遗传信息交换的方法。大肠杆菌和酵母是最常用来做体内同源重组生物“细胞工厂”,用大肠杆菌中的Red/Rec重组系统拼接较大的chunks/megachunksDNA分子构建和用酵母的TAR重组系统拼接成megachunks/最终构建都非常有效。但在酵母中的重组效率要比在大肠杆菌中的更高。一般情况下,质粒在酵母中是单拷贝,需要将大构建转化到大肠杆菌中进行质粒制备。
将有同源重组臂的目的片段和载体同时转入酵母细胞,在体内进行同源重组的技术被称为TAR技术,但由于这种TAR技术目前还并未被广泛的开发利用,所以要得到更高的重组效率,还有许多要改进和需要注意的地方。如重组臂不能与酵母基因组有同源,以免转入的序列与酵母基因组发生重组;重组臂的长度应该根据目的片段的大小而有所不同;大分子DNA片段产物的浓度要足够高,才能得到更多的阳性克隆,否则仅仅是筛选到阳性克隆就要花费大量的时间,GENEWIZ优化后的大分子DNA回收方法,比一般的方法回收效率可以提高2-3倍;酵母的转化效率,也是TAR技术中一个至关重要的环节。此外根据目前的研究发现,从酵母中制备的大分子质粒,很大一部分都不是完整的环状质粒DNA,从而导致了在下一步转到大肠杆菌的时候阳性菌落较少。GENEWIZ采用了优化的酵母质粒制备方案,可以从酵母中制备更加完整的大分子质粒,在转化大肠杆菌时也有更高的阳性克隆率。当然有些对酵母或大肠杆菌有毒性的基因,构建都会更加困难。
Reference
[1]https://www.cbinsights.com/research/industrial-synthetic-biology-market-map/
[2]Hughes R A, Ellington A D. Synthetic DNA synthesis and assembly: putting the synthetic in synthetic biology [J]. Cold Spring Harbor perspectives in biology, 2017, 9(1): a023812.
[3]Kwok R. Five hard truths for synthetic biology: can engineering approaches tame the complexity of living systems? Roberta Kwok explores five challenges for the field and how they might be resolved [J]. Nature, 2010, 463(7279): 288-291.
[4]Voigt A C. Genetic parts to program bacteria [J]. Current Opinion in Biotechnology, 2006, 17 (5): 548-557.
[5]Ro DK, Paradise EM, Ouellet M, et al. Production of the antimalarial drug precursor artemisinic acid in engineered yeast. Nature. 2006,13(440): 940-943.
[6]Meng F, Ellis T. The second decade of synthetic biology: 2010–2020[J]. Nature Communications, 2020, 11(1): 1-4.
[7]Gibson D G, Glass J I, Lartigue C, et al. Creation of a bacterial cell controlled by a chemically synthesized genome[J]. Science, 2010, 329(5987): 52-56.
[8]Hutchison C A, Chuang R Y, Noskov V N, et al. Design and synthesis of a minimal bacterial genome[J]. Science, 2016, 351(6280).
[9]Richardson S M, Mitchell L A, Stracquadanio G, et al. Design of a synthetic yeast genome [J]. Science, 2017, 355(6329): 1040-1044.
[10]Xie Z X, Li B Z, Mitchell L A, et al. “Perfect” designer chromosome V and behavior of a ring derivative[J]. Science, 2017, 355(6329).
[11]Wu Y, Li B Z, Zhao M, et al. Bug mapping and fitness testing of chemically synthesized chromosome X [J]. Science, 2017, 355(6329).
