CRISPR技术新的突破与展望——GBE碱基编辑技术

前言 ·2020年诺贝尔化学奖颁发给了法国和美国科学家Emmanuelle Charpentier、JenniferA.Doudna,以表彰她们“开发出一种基因组编辑方法”-即CRISPR/Cas9基因编辑技术。CRISPR/Cas9基因剪刀其实大家已经非常熟悉,而在CRISPR/Cas9技术基础上开发的单碱基编辑技术——GBE系统则是CRISPR技术新的突破,代表着未来的方向。
今年7月在Nature Biotechnology杂志背靠背发表的两篇关于CRISPR技术的新文章(Kurtet al., 2020; Zhao et al.,2020),为我们揭示了一项全新的碱基编辑技术——GBE,可以将C碱基高效转变为G碱基,进一步拓宽了碱基编辑技术的适用范围,为碱基编辑技术在基础科研和临床治疗上的应用提供了新的助力。不仅如此,GBE有别于CBE和ABE的编辑方式,也为碱基编辑提供了一种全新的设计思路,让我们朝着碱基自由变换的终极目标继续迈进。

1. 碱基编辑技术
碱基编辑技术诞生于2016年,由DavidR. Liu实验室首先提出(Komoret al., 2016; Gaudelli et al.,2017),可以细分为CBE与ABE技术。两者都是依托于CRISPR的DNA定位能力,将C碱基脱氨酶或A碱基脱氨酶定位到基因组的特定位置,催化特定位置的C或A进行脱氨反应,转变为U或I,然后在DNA复制的过程中被当作T或G,实现C到T或A到G的转换(图1)。借助CRISPR的DNA精准定位能力以及脱氨酶的高效催化能力,而又不依赖于同源重组这样低效率的DNA修复过程,碱基编辑技术可以实现到目前为止最为高效的基因组精准编辑。编辑效率和准确性在要求严格的临床治疗中尤为重要,所以碱基编辑技术在临床转化中极具潜力,而限制其发展的首要问题则是如何实现其他碱基的转换过程。
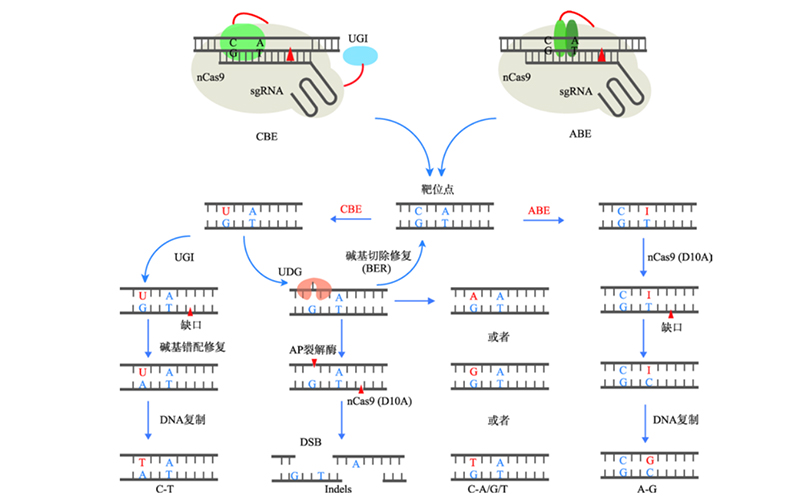
图1 CBE 和 ABE工作模式图(Zongand Gao, 2019)
NatureBiotechnology上发表的这两篇文章标志着GBE技术的诞生,也进一步扩充了碱基编辑技术的武器库。其中一篇文章是由中国科学院天津工业生物技术研究所张学礼和毕昌昊实验室合作完成(Zhaoet al.,2020),作者在其文章中详细描述了GBE技术的发现和发展过程。值得介绍的是,本文中的NGS测序是根据金唯智的建库方案并在金唯智完成的测序。
2. GBE的契机
GBE简单来说,可以理解为CBE技术的副产物。在CBE的过程中,C变为U后,会被细胞內源的UNG(Uracil-DNAglycosylase,UDG)酶去除U碱基,变为所谓的AP状态(apurinic/apyrimidinic,意思为无嘌呤无嘧啶),进而引发碱基切除修复(base-excisionrepair,BER),按互补链的G重新加上C;或者被AP裂解酶(APlyase)识别,引发DNA双链断裂,造成随机突变。但不论哪一种情况发生,都是在降低CBE技术的效率,并且可能引发随机突变,所以在CBE技术的发展过程中,引入了外源的UGI(uracilglycosylaseinhibitor)抑制UNG的活性,保护U碱基,提高C到U再到T的转换效率。
而本文作者和之前的一些研究者都发现了碱基处在AP状态下存在的一个特殊的现象,缺失的碱基可能被修复为其他三种碱基(图2),这个现象曾经被当成一种不可控的副作用,通过UGI的作用基本能够避免。而本文作者却另辟蹊径,将这个现象作为一个突破口,寻找将C转化为A或G的可能性。
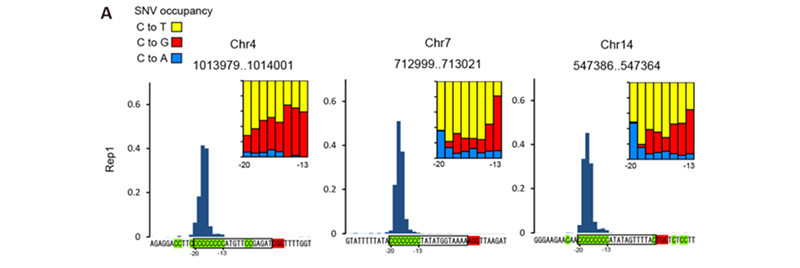
图2 CBE(nCas9D10A-PmCDA1)引发的碱基突变(Nishidaet al., 2016)
作者首先单纯测试了胞嘧啶脱氨酶AID在大肠杆菌中的效果,发现C脱氨后基本只会变成T或者A(图3左上)。T是C脱氨后变为U的最终产物,而A自然就是U被去除后产生的现象。而之所以只出现A碱基,可能是细胞内某种未知的修复机制修复AP所导致,至少作者给出了两个现象支持了这种观点:在Eco.li中敲除UNG,U不会被去除,就没有再出现C转换为A的现象(图3左下);或者使用dCas9替代nCas9,C转换为A也不会发生,这点明显不同于CBE,CBE使用dCas9或者nCas9都能催化C到T的过程(图3右下)。作者接下来的思路就是能否抑制C到T的过程,只保留C到A的过程,这样我们就获得了一种新的碱基编辑方式。为抑制C到T这个过程,作者选择增强去除U碱基的这个过程,在nCas9上再融合一个UNG,在原位对AID脱氨产生的U进行去除。测试的效果也比较理想,基本抑制了C到T这个过程,C到A变成了主要的碱基编辑方式(图3右上)。

图3 在大肠杆菌中nCas9-AID(左上)、Ung-nCas9-AID(右上)、nCas9-AID与Ung敲除(左下)、dCas9-AID(右下)的碱基编辑情况(Zhaoet al., 2020)
但当作者在哺乳动物细胞HEK293T中进行相同的实验时,发生的现象却并不相同,细胞中修复AP的偏好变成了G碱基,融合UNG后,也能够抑制C到T的过程,C到G成为主要的碱基编辑方式(图4),作者便将这种新技术称为GBE。相比于它的前辈CBE和ABE,GBE不能算是一种典型的碱基编辑方法:
首先在大肠杆菌和HEK293T中不同的修复偏好,很有可能由不同的修复机制导致的,该技术的普遍适用性和稳定性还有待检测;
其次在HEK293T中的实验数据也显示GBE的编辑窗口和编辑效率都不如CBE和ABE,相比于CBE和ABE完全是利用高效的酶催化反应,GBE背后未知的修复方式还需要很多研究和优化。
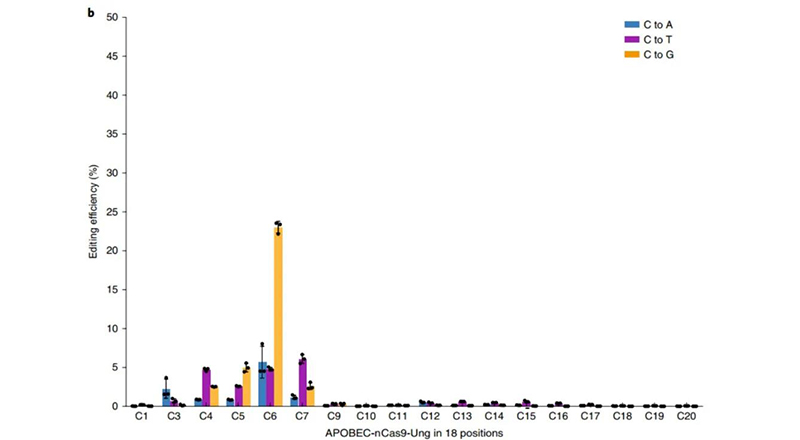
图4 在HEK293T中APOBEC-nCas9-UNG的碱基编辑情况(Zhaoet al., 2020)
长期以来,基因的精准编辑都需要借助同源重组(HDR)完成,但是HDR在很多细胞中编辑效率极低。同时,大部分HDR需要借助Cas9产生DNA双链断裂来提升效率,不可避免的非同源末端连接(NHEJ)会导致随机突变。HDR的这两个缺点严重限制了基因精准编辑领域的发展。由于HDR和NEHJ都是细胞内源的DNA修复机制,不便进行人为改造,基因精准编辑的另一个流派就是不利用Cas9造成的DNA双链断裂,也就不会引发NHEJ导致的随机突变,利用全新的修复机制进行精准编辑,碱基编辑和先导编辑(primeediting)技术就是其中的佼佼者。
3. 碱基编辑未来展望
碱基编辑作为以超高效率而闻名的基因精准编辑技术,GBE的出现进一步提高了它的适用范围,也再次为我们阐述了碱基编辑技术未来的发展方向:
1、首先,CRISPR的识别序列覆盖度在不断的改造中已经越来越高,最新的改造型SpRY-Cas9已经基本突破了PAM序列的限制,理论上可以识别所有序列。在未来,序列识别将不受限制,碱基编辑才能够真正覆盖到全基因组上。本文的作者也在大肠杆菌中尝试了类似的工作,当能够任意靶向一个碱基或者与它互补的碱基时,借助GBE在大肠杆菌中引发C到A,碱基编辑技术可以将C/G或者A/T编辑为任意一个其他的碱基(图5),实现全碱基编辑(NBE)技术。
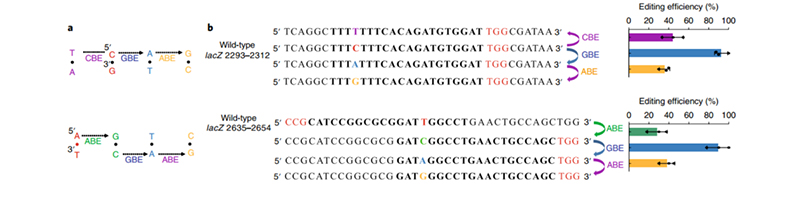
图5 大肠杆菌中的NBE技术展示(Zhaoet al., 2020)
2、高效率一直是碱基编辑技术的最大优势,从原理上避开了低效率的同源重组以及不稳定的非同源末端连接,该技术未来的发展重点一定会围绕寻找新的高效编辑酶,实现更多的碱基删除、添加或者转换过程,在保证酶高效催化活性的前提下,实现更多的碱基变换过程。
3、碱基编辑的编辑窗口一直是该技术发展的一个痛点,由于Cas9/sgRNA与DNA形成R-loop时,非靶向的DNA单链不被Cas9包裹,处于游离状态,碱基编辑的编辑窗口一般都是很宽泛的6到7个碱基,并且目前为止都没有很好的办法缩小编辑窗口。在这个方面,GBE较为单一的编辑窗口(集中在第6个碱基上)反而成为了一种优势,配合全序列识别,能够做到较为精准的基因编辑。
4. 小结
CRISPR技术已经成为每个生物实验室必备的基因编辑工具,同时也在积极向临床医疗领域转化,形成一个潜力巨大的市场。在医疗领域,安全性永远被放在第一位,这就要求CRISPR能够实现准确而高效基因编辑,像碱基编辑这样的CRISPR新技术也就是在近几年应运而生,并在短时间内取得了巨大的进步,为基因编辑这个生物医疗的大方向提供强劲持续的动力。
参考文献: [1]Gaudelli,N.M., Komor, A.C., Rees, H.A., Packer, M.S., Badran, A.H., Bryson,D.I., and Liu, D.R. (2017). Programmable base editing of A*T to G*Cin genomic DNA without DNA cleavage. Nature 551, 464-471. [2]Komor,A.C., Kim, Y.B., Packer, M.S., Zuris, J.A., and Liu, D.R. (2016).Programmable editing of a target base in genomic DNA withoutdouble-stranded DNA cleavage. Nature 533, 420-424. [3]Kurt,I.C., Zhou, R., Iyer, S., Garcia, S.P., Miller, B.R., Langner, L.M.,Grunewald, J., and Joung, J.K. (2020). CRISPR C-to-G base editors forinducing targeted DNA transversions in human cells. Nat Biotechnol. [4]Nishida,K., Arazoe, T., Yachie, N., Banno, S., Kakimoto, M., Tabata, M.,Mochizuki, M., Miyabe, A., Araki, M., Hara, K.Y., et al.(2016). Targeted nucleotide editing using hybrid prokaryotic andvertebrate adaptive immune systems. Science 353. [5]Zhao,D., Li, J., Li, S., Xin, X., Hu, M., Price, M.A., Rosser, S.J., Bi,C., and Zhang, X. (2020). Glycosylase base editors enableC-to-A and C-to-G base changes. Nat Biotechnol. [6]Zong,Y., and Gao, C.X. (2019). [Progress on base editing systems]. Yichuan = Hereditas 41, 777-800.
