金唯智新型技术测通AAV-ITR区域
腺相关病毒基因组(adeno-associated virus, AAV)的反向末端重复序列(inverted terminal repeat, ITR)能形成高度稳定的二级结构,利用现有技术很难进行序列验证。为了突破这重障碍,GENEWIZ研发了一种自主知识产权技术,运用原创的测序方法,清晰地分析这些复杂序列,助力研究人员有效地评估AAV-ITR区域的完整性。
背景介绍
腺相关病毒(AAV)是一种单链DNA病毒,在基因治疗中通常作为载体将外源基因转入患者中。AAV基因组的每条单链DNA由4680个碱基构成,包括两端145bp的反向末端重复序列(ITR)和中间的2个开放性阅读框。ITR在病毒的复制和包装过程中起着关键作用,并且参与了病毒基因组在宿主基因组上的整合和逃逸过程。该序列中CG含量达80%以上,其前125bp(1~125)序列依次可分为A、B、B’、C’、C、A’等不同的区段(图1),其中B与B’、C与C’反向互补,可形成T型发夹结构,作为AAV的DNA自我复制的起点,随后的20bp形成特有的D序列。

图1:AAV2的ITR序列的示意图
ITR由两个臂回文(B-B’和C-C’)和一个长茎回文(A-A’)组成。D序列在AAV基因组的每一端只出现一次。
为产生重组腺相关病毒(rAAV),哺乳动物或昆虫细胞通常需要共转染两个或者三个质粒——其中一个或两个质粒提供AAV包装复制元件和腺病毒辅助元件,另外一个质粒(AAV质粒)包含两端带有ITR序列的目的基因表达框。AAV质粒中ITR区域的完整性对rAAV的产生至关重要。缺失突变的ITRs会降低完整AAV病毒的产量,增加非目的DNA包装的产生1。然而,在使用大肠杆菌进行质粒扩增过程中,AAV质粒的ITR区域经常发生突变。研究表明,ITR序列的不同区段缺失,都会对rAAV的产生造成影响:
A-A’ region
A-A’ region 中的串联重复序列5’-GAGC GAGC GAGC GCGC-3’是 A-A’ region 甚至整个ITR中的核心序列,其最明确的功能是与 Rep 蛋白结合,因此被称为 Rep 结合位点(Rep Binding Element, RBE)。RBE 涉及 AAV 基因组的复制、转录及前病毒的整合,几乎所有 ITR的重要功能都是由Rep与RBE结合所起始的。
当突变RBE序列中的第 2、3个GAGC或只突变第 3个GAGC时,AAV基因组DNA的复制水平显著下降2。
A-A’ region 中还含有特异性的TRS序列(terminal resolution site),即 5’-TTGGCCAC-3’。ITR中的TRS序列参与AAV的拯救和复制途径。
删除 A-A’ region 中的非 RBE 序列,只保留 RBE 及附近的部分序列时,Rep 对此突变 ITR 的亲和力下降至少 10 倍3。
B-B’及 C-C’ regions
wtITR 中存在两个小回文结构:B-B’及C-C’ regions。这两个回文对Rep的结合也有一定作用。
位于 B-B’ region 中的被称为 RBE’的序列(5’-CTTTG-3’)与 Rep有较强的亲和性。如果CTTTG序列中的后两个T发生突变,则严重影响ITR与Rep的结合,进而影响AAV的产生4。
D region
ITR 中 A-A’、B-B’及 C-C’ regions 均形成回文双链结构,只有D region为单链。有研究发现,D region缺失,即AAV载体质粒中两个ITR均不含D region时,病毒基因组不能被包装。说明D-region在AAV病毒的包装中起关键作用5。
因此,对AAV质粒的ITR序列进行准确的分析将会对rAAV的质控具有重要的意义。
面临挑战
以目前的技术,可以通过限制性内切酶的酶切反应来研究ITR的定位及其完整性。但凝胶电泳的分辨率较低,很难检测到点突变或小缺失。
直接对ITR区域进行测序可以提供足够的分辨率,但是常规Sanger测序试剂盒中的聚合酶会被高GC含量和较长的回文序列(>100 bp)折叠成T型发夹结构阻碍,导致测序失败。其难点在于两个方面:1. 测序酶不同于野生型的聚合酶,它是经过突变的高温聚合酶,该聚合酶的持续DNA合成能力被降低且易被发夹结构特别是极其稳定的ITRs所抑制;2. 这些常规试剂盒中使用了脱氧核糖核苷三磷酸类似物,以达到更高的毛细管电泳峰分辨率。然而在复杂结构特别是发夹结构中,脱氧核苷三磷酸类似物并不能有效地结合DNA聚合酶。
由于二级结构在线性模板中不稳定,所以通过PCR扩增ITR区域,然后对扩增产物进行测序可以得到更好的结果6。然而,这种两步法的测序需要特殊的引物设计和循环设置。并且这种检测方法对于ITR区域的两端的序列要求较高,成功率也不能保证。而且,每一条新构建的AAV质粒均含有两个ITR序列,由于PCR反应是非通用的,因此针对两个ITR区域需要设计两种PCR反应。
解决方案
为了解决这一难题,GENEWIZ开发了一种新型的AAV-ITR测序方法,可以改善ITR区域的测序信号并延长读取长度。
为了验证我们的ITR测序方法,将其与标准方法进行比较,我们首先使用BigDye®cycle sequencing kit(Thermo Fisher)对含有AAV2 ITRs的质粒进行测序。不出所料,在ITR发夹结构开始处,测序信号突然下降(图2),导致测序过早终止。添加常见的添加剂,如DMSO和/或甜菜碱也不能改善这一结果(数据未显示)。
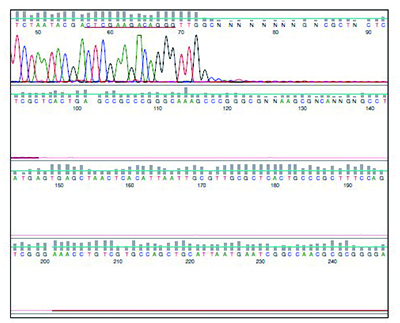
图2:使用BigDye®cycle sequencing kit对AAV2的ITR区域进行测序
红色下划线标记序列后的碱基“TTGGC”是一个无法成功测序的125 bp ITR发夹结构的开端。
而GENEWIZ的新型AAV-ITR测序方法可以测通ITR发夹结构并且在整个ITR区域没有明显的信号损失,达到了与常规测序反应类似的读取长度(图3)。此外,我们还研究了引物结合位点对ITR测序质量的影响。我们发现,当引物与ITR区域之间的距离为150-350bp时,通常可以获得最理想的测序质量(数据未显示)。
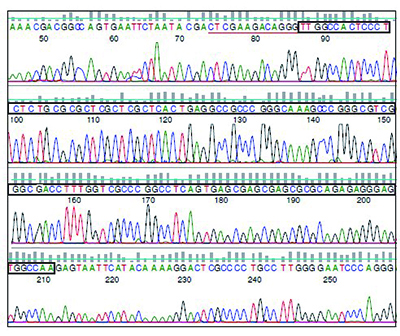
图3:使用GENEWIZ ITR测序法测定AAV2的ITR区域的色谱图
红色下划线的序列表示图2中提到的相同序列。黑框内显示完整的125bp ITR发夹结构。
rAAV质粒中ITR完整性的定性评价
目前,rAAV质粒主要在细菌中扩增。然而,由于细菌中同源重组系统,ITRs经常发生重排。细菌繁殖后,rAAV质粒中最常见的突变之一是ITR的B-B’或C-C’臂缺失(图4)。这种缺失使T型ITR发夹的茎更长,比野生型ITR更加稳定。在对数百条来自不同细菌细胞系中扩增的rAAV质粒进行测序后,我们发现,我们发现,GENEWIZ ITR测序方法可以测通野生型ITR序列(图4和图5A)以及更有挑战性的缺失突变的ITR序列(图5B),并且能帮助定性评估ITR区域甚至是缺失突变ITR区域的完整性。
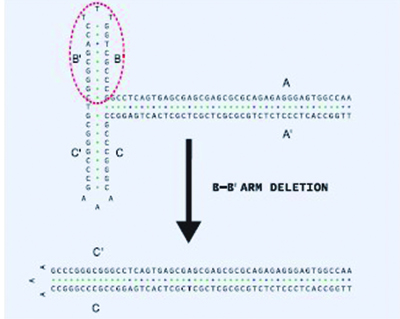
图4:AAV2的ITR区域中B-B’臂缺失的示意图
野生型AAV2的ITR区域是一个125bp的T型发夹结构,而缺失突变的ITR是一个103bp的发夹结构,其茎部更长。
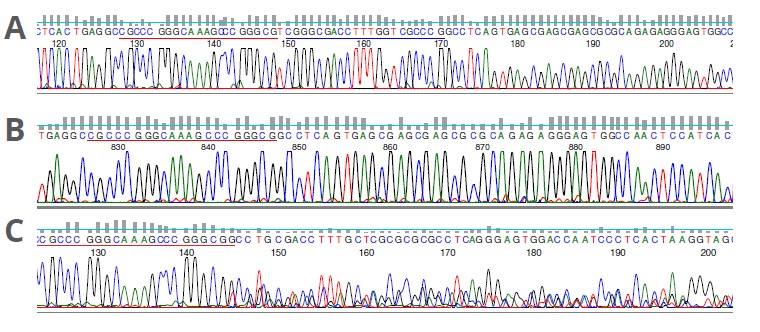
图5:利用GENEWIZ ITR测序方法分析rAAV质粒中ITRs的完整性
A) GENEWIZ AAV-ITR测序法测定野生型AAV2 的ITR区域的色谱图。C-C’臂和B-B’臂分别用红色和蓝色标出。B) GENEWIZ AAV-ITR测序法对AAV2的缺
失
突变ITR区域进行色谱分析。测序结果显示B-B’臂出现缺失。C) GENEWIZ AAV-ITR测序法测定缺失突变的AAV2 ITR区域的色谱图。测序结果表明,
该质粒是完整ITR和缺失突变ITR的混合物。在155-157bp处的TTT峰与完整的ITR序列一致,其中存在缺少B-B’臂的质粒模板。
GENEWIZ AAV-ITR测序方法的优化
一些rAAV质粒在ITR区域的5’端前部含有一个同聚物G区域(图6A)。现有的Sanger测序方法测通同聚物G区域非常困难,GENEWIZ的AAV-ITR测序方法也面临同样的困难。伴随着Poly-G的测序过程,测序信号值也逐渐降低,并导致未测序至ITR发夹结构测序反应就提前终止 (图6B)。目前尚未有合适的方法能解决poly-G区域对ITR发夹结构造成的热力学障碍。但是,GENEWIZ的AAV-ITR测序方法能通过反向测通ITR发夹结构和poly-C重复区域(图6A),进而提高该区域的数据质量及读取长度(图6C)。

图6:GENEWIZ的ITR测序法对含有poly-G序列的AAV质粒进行测序
A) ITR区域的5’端前部含有一个同聚物G区域的示意图;黑色和绿色箭头分别表示正向测序和反向测序。B)采用GENEWIZ的AAV-ITR测序
法,正向对同聚物G + ITR区域进行测序的色谱图。C)采用GENEWIZ的AAV-ITR测序法,反向对同聚物G + ITR区域进行测序的色谱图。
GENEWIZ的AAV-ITR测序方法能有效评估AAV质粒中ITR区域的完整性。它可以迅速准确地检测到ITRs区域的突变,为下游故障的排除节省时间和精力。我们选择了AAV2的ITR区域进行测试,因为AAV2是最典型的、最常用的AAV血清型。AAV不同血清型含有相似的ITR序列,目前我们正在对不同AAV血清型的ITR区域进行测序以及包括其他的DNA病毒等。这种AAV-ITR区域的测序方法可以广泛应用于热力学稳定的发夹结构的测序。

金唯智美国客户反馈
订购流程:
1. 下载并填写《金唯智ITR测序送样单》并发送至邮箱:Support.Asia@Azenta.com
2. 将样品按送样要求寄送或交给取样员
3. 等待AAV-ITR测序结果(3-5个工作日)
4. 完成测序
References
1. Savy A. et al., Impact of inverted terminal repeat integrity on rAAV8 production using the baculovirus/Sf9 cells system. Hum. Gene Ther. Methods. 2017:28(5):277-289.
2. Bishop, B. M., et al. “Role of the terminal repeat GAGC trimer, the major Rep78 binding site, in adeno‐associated virus DNA replication.” FEBS letters 397.1 (1996): 97-100.
3. Im, Dong-Soo, and Nicholas Muzyczka. “The AAV origin binding protein Rep68 is an ATP-dependent site-specific endonuclease with DNA helicase activity.” Cell 61.3 (1990): 447-457.
4. Ryan, John H., Sergei Zolotukhin, and Nicholas Muzyczka. “Sequence requirements for binding of Rep68 to the adeno-associated virus terminal repeats.” Journal of virology 70.3 (1996): 1542-1553.
5. Bleker, Svenja, Florian Sonntag, and Jürgen A. Kleinschmidt. “Mutational analysis of narrow pores at the fivefold symmetry axes of adeno-associated virus type 2 capsids reveals a dual role in genome packaging and activation of phospholipase A2 activity.” Journal of virology 79.4 (2005): 2528-2540.
6. Kieleczawa J. Fundamentals of sequencing of difficult templates–an overview. J Biomol. Tech. 2006:17(3)207-17.
7. Grimm D. et al., Liver transduction with recombinant adeno-associated virus is primarily restricted by capsid serotype not vector genotype. J. Virology. 2006:80:426-439.
