Plasmid-EZ 测序常见问题
1. Plasmid-EZ全质粒测序原理是什么?
金唯智全质粒测序使用的是Oxford Nanopore Technology (ONT)纳米孔测序。Nanopore序列测定依赖于电流信号到碱基序列信号的转换。其核心是让单链核酸分子在马达蛋白驱动下穿过纳米孔,不同碱基(A、T、C、G 等)因结构差异,通过时会导致跨膜电流发生特征性变化。电极实时捕获这些电流信号,再经算法分析信号的幅度、持续时间等特征,推断出碱基类型。该技术无需 PCR 扩增,可直接实现单分子实时测序。
2. 质粒测序结果的准确性如何?
Plasmid-EZ使用ONT平台最新R10测序芯片与kit14试剂,原始测序准确度为99.5%,经一致性分析单分子准确性可提升至99.9%。
通过我们对测序数据进一步分析组装矫正得到一个高质量的一致性序列,准确度大大提升,与Sanger测序确认正确的参考序列比对,序列一致性高于99.9972%。
3. Plasmid-EZ测序有何优势?
- 最快1天可出结果,快速高效
- 不需要提供已知序列信息或测序引物
- 样本通量可灵活调整
- 测序对质粒大小、结构复杂度(高GC、ITR序列等)无限制
- 为质粒质检提供更全面的信息
- 质粒结构具有高准确度
4. Plasmid-EZ与Sanger测序对比有何不同?
| 特点 | Sanger 测序 | Plasmid-EZ |
|---|---|---|
| 测序区域 | 一般仅针对目标基因,可通过 Primer walking 多反应测序拼接获得完整质粒 | 可直接测完整质粒 |
| 引物要求 | 是 | 否 |
| PCR 要求 | 是 | 否 |
| 通量 | 低 | 高 |
| 识别插入元件的方向 | 无法识别方向 | 是 |
| 解析结构复杂区域 | 否 | 是 |
| 测出多聚体质粒 | 否 | 是 |
| 测出混合质粒或杂质 DNA | 否 | 是 |
| 周期 | 以 4 个反应为例:3-4 天 | 最快 1 天 |
5. Plasmid-EZ项目的送样要求是什么?
环状质粒
- 质粒单一,无基因组污染或降解(推荐使用Qubit定量,避免浓度虚高)
- 片段≤10Kb:浓度≥20ng/μl,体积≥20μl;
- 片段10Kb-20Kb之间:浓度≥30ng/μl,体积≥20μl;
- 片段≥20Kb:浓度≥40ng/μl,体积≥20μl;
- 纯度要求:OD260/280为1.8-2.0,OD260/230≥2.0。
菌液/平板菌落(片段≤20Kb)
- 菌液:提供体积≥200μl新鲜菌液(OD>0.8),装于1.5ml离心管中封口保存;
- 直抽菌液:过夜培养的新鲜大肠杆菌菌液2ml或2ml菌液的沉淀;
- 平板菌落:单菌落,无明显卫星菌落,需将待测的菌落圈选标注并编号。
6. 为什么我应该对整个质粒进行测序,而不是只测我感兴趣的区域?
仅测感兴趣的区域,无法排查载体骨架区域的潜在错误(碱基突变/插入/缺失),进而导致耽误实验进程。质粒插入片段正变得越来越长、越来越复杂,牛津纳米孔技术(ONT)测序成本低且非常适合解决长片段和难度的重复序列测序。Sanger测序无法确认质粒是否为多聚体,ONT能帮忙确定您的质粒是否为多聚体或混合质粒,避免质粒误用造成实验成本的额外浪费。
7. Plasmid-EZ测序周期是多久?
最快1天反馈结果,各实验室上机时间如下:
- 苏州实验室上机时间:周一 ~ 周五
- 天津实验室上机时间:周一 ~ 周六
- 广州实验室上机时间:周二 ~ 周六
8. Plasmid-EZ结果保存时间是多久?
原始数据自发货之日我们免费为您保存1个月,超期数据将默认被清理。收到数据后,请您及时查看和下载,避免耽误您的实验进程。3个月内的超期数据,您可以联系技术支持尝试线下付费查找(收费100元/份订单)。
9. Plasmid-EZ如何快速入门?
您可以查阅金唯智的Plasmid-EZ快速入门指南链接。
10. 突变检测能以Plasmid-EZ结果为准吗?
由于纳米孔测序原始测序读长存在一定比例的随机突变,提供的突变信息为参考值,具体变异建议以Sanger或NGS验证结果为准。
11. 若样品为混合质粒,如何查看多个组装结果?
组装结果也有可能包含多个序列,如果出现这种情况,01_Assembly 文件夹中的 genome.fa 文件将包含所有组装好的结果序列。
在SnapGene Viewer中打开此文件,您将看到所有结果序列的列表,以及来自组装程序的一些详细信息,如下图,”circular”表示正常组装为环形序列,“linear”表示线性序列。

例如,若组装得到了两个结果序列:一个是完整的环状质粒,另一个并非完整环状序列。那么,第二个结果序列很可能是组装过程中产生的组装伪像,而非真正的变异。
相反,如果看到多个环状组装序列,且原始数据长度分布中能看到对应各自长度的数据峰,则这可能代表样本中存在多个质粒。
如果所有结果序列都只有少量reads,则可能表明样本的组装质量较低或样品本身数据量较少。
12. 成功的结果reads分布图是什么样的?
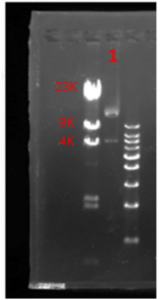
正常样本的reads分布图以及胶图如下,可以看到reads分布有明显的主峰,短片段比例较低,胶图条带清晰,供参考。
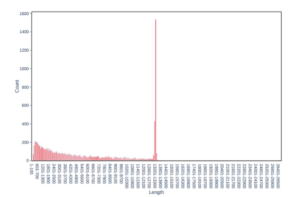
正常样本reads长度分布图
正常样本胶图
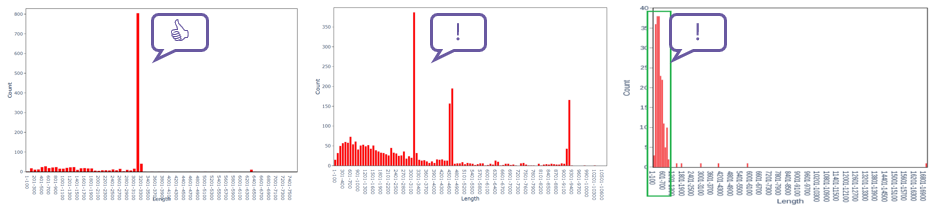
13. 为什么我的样品组装失败了?
若样本组装失败,Result文件夹中将仅包含00_QC文件夹。文件夹内包含原始数据、样本读长、数据质量等相关信息。


组装失败的最常见原因是样本浓度未达到所需的送样要求,或存在样品浓度虚高问题。浓度过低可能导致建库过程中片段化加剧及/或样本测序读长(reads)数量不足。我们强烈建议您在送样前使用Qubit或等效仪器检测样本浓度,以降低失败风险。
从测序读长长度分布图可一定程度上解释出现不同组装结果的原因:左图测序读长峰形单一,表明样本为单克隆质粒,通常可成功组装;中图出现多个峰,说明样本中可能存在片段缺失、重组或多聚体质粒等情况,可能出现多条组装结果;右图测序读长以短片段为主,不存在主峰,说明样本中可能存在严重降解或浓度虚高等情况,高概率会导致组装失败。
14. Plasmid-EZ收费如何?
Plasmid-EZ无论测序成功或失败均会正常收费。为了提高测序成功率,建议您送样前对质粒进行凝胶电泳,预先排除降解或浓度虚高问题。
15. 结果文件格式及打开方式是什么?
FASTA(fa)格式,http://en.wikipedia.org/wiki/FASTA_format,可以用记事本或其他文本编辑器直接打开。
gbk格式文件,可以在SnapGene Viewer软件中打开。
结果文件中统计表格均为制表符(Tab)分割的文本,Windows用户建议使用excel或openoffice等办公软件以表格形式打开。
Unix/Linux用户可以使用more或less命令查看。
16. 样本浓度低于送样要求怎么办?
浓度低的样品可能会造成在混样中占比过低导致测序覆盖深度低甚至组装失败。
17. Plasmid-EZ服务是否可以提交混合质粒样本进行测序?
该服务适用于单克隆质粒样本,也可以检测混合质粒样本。但如果提交混合质粒样本,则无法保证测序和分析结果。
(1) 如果样本中含有相似度非常高的克隆,比如载体相同,会导致组装过程中部分reads无法区分,组装不完整或错误;
(2) 如果目标克隆在混样中占比不高,可能会导致目标克隆数据占比过低,覆盖深度不够从而组装失败。
18. Plasmid-EZ项目交付哪些结果文件?
a) 原始测序数据(Report/Result/00_QC /raw.fastq文件)
b) 可交互分析结果报告(Report/Report.html文件)
c) 可交互分析测序reads长度分布图(Report/Result/00_QC/LengthDist.html文件)
d) 可交互分析测序reads质量分布图(Report/Result/00_QC/Qual.html文件)
e) 质粒组装结果序列(Report/Result/01_Assembly/genome.fa文件,我们建议将此结果导入snapgene中进行查看)
f) 测序数据比对质粒基因组组装序列bam文件(Report/Result/01_Assembly/bam文件)
g) 质粒基因组各位置的测序碱基组成统计文件(Report/Result/01_Assembly/Base-composition.xls文件)
h) 质粒图谱含结构注释(Report/Result/02_Annotation/*_pLann.html文件)
i) 质粒基因组组装序列及结构注释GenBank文件(Report/Result/02_Annotation/*_pLann.gbk文件)
19. 如何处理与参考序列不一致的位置?
纳米孔测序易在甲基化修饰位点(特别是GATC序列),同聚物区域(poly区)出错或产生InDel。
对于poly区域,建议使用IGV软件查看测序数据比对质粒基因组组装序列的bam文件,或者直接查看Base-composition.xls文件,判断是否存在reads符合参考序列poly区域长度。若存在,则建议以参考序列为准。若参考序列中存在超长的poly区域,则建议使用Sanger或NGS等多种平台进行验证。
对于非poly区域出现的与参考序列不一致的位点,建议查看Base-composition.xls文件,建议根据reads数进行分析确定。
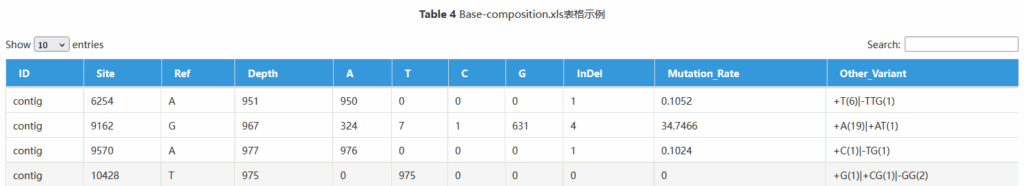
列名解释:
(1) ID:拼接序列ID;
(2) Site:拼接序列上的位置;
(3) Ref:拼接序列在该位置上的碱基;
(4) Depth:拼接序列在该位置的覆盖深度;
(5) A/T/C/G:支持该处为A/T/C/G碱基的reads数量;
(6) InDel:支持该处为单碱基缺失的reads数量;
(7) Mutation_Rate:拼接序列在该位置处的突变频率(%)-> (覆盖深度-支持原有碱基的reads数)*100/覆盖深度;
(8) Other_Variant:其他突变类型(包括插入以及大于1bp的缺失,例如”+T(3)”则代表3条reads支持在该位置存在一个T碱基的插入)。
20. 拼接序列是环状序列还是线性序列?
质粒基因组组装结果genome.fa文件由若干条contig序列组成,每条contig序列为1条拼接结果,若contig后缀为circular,则表明该条序列为环状序列;若contig后缀为linear,则表明该条序列为线性序列。
21. 什么会导致碱基识别错误或可信度低?
牛津纳米孔技术最常见的错误模式包括:同聚物片段(尤其是长度>8 bp的)中的缺失、Dam甲基化位点GATC处的错误,以及Dcm甲基化位点CCTGG或CCAGG中间位置的错误。随着牛津纳米孔技术(ONT)测序化学试剂和碱基识别软件的未来更新,这些局限性有望得到改善。
22. 每个质粒的测序覆盖度是多少?
测序覆盖度高度依赖于最终用户在提交样本之前确定的样本质量,因此无法保证。正确提交的质量良好的样本通常会产生数千条的测序reads。
23. Plasmid-EZ可以检测到质粒多聚体吗?
Plasmid-EZ服务正是检测质粒多聚体样本的有效方法,它可以产生较长的读长,也可以检测质粒内的大片的重复序列。通过查看原始数据长度分布信息可清晰了解质粒的聚体情况。
如果样本中存在比例较高的多聚体,可能会影响组装结果。
